2009/04 |
2009/05
|
2009/06
1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
2009年5月 3日
 チャリ x フェリー
チャリ x フェリー
本日は連休中のメインイベントなのです。

待ち合わせに指定した明石駅に着いたのが早かったので、外に出て池を見てたんですが。岸に近いところでぽつぽつと黒い点がたくさん水面に出ててちょっと不思議空間やった。何かというと亀の頭なんですがね。なんというか、多い。ならにも同じような池に亀がたくさんいたところがあったけど、そこは岩とか木が出ててそこに固まって日向ぼっこしてたのが、ここじゃ頭だけ。
ぼーっとしてたら突然小さい女の子に挨拶された。…これもまた謎。
11時過ぎに全員集まって、まぁ早速ご飯を食べに行くことにして、その前にチャリを借りると。
JR西日本のレンタサイクル「駅リンくん」で自転車借りて淡路島へ!!
まぁ↑でやってることをそのままなぞっただけです。もともとは明石で鯛めしを食べたいだけやったんやけど…。それ以外にすることがほとんど見つからなかったので、チャリで淡路島に行ってみるのもいいかと思いついてしまっただけで。可能性の検証に数日もかけた(というか、思いついたのも1週間前を切ってから)。
たこフェリーの乗り場の食事どころで鯛めしは軽くクリアできることが分かったので、午後はゆっくり淡路島…多分大丈夫だろうと。野島断層を見るくらいの距離なら、いくら運動不足といっても往復するくらい4時間あれば。

震災の壁(神戸の壁)です。唐突に。この時点で14時半を回ってるんですが、ここに来るまでにひたすらチャリで海岸沿いの道路を走ってきただけ。船に乗ってる間は外にも出ず(風が強かったし)、明石海峡大橋の上が混んでるのを横目に6人で喋りながらさくっと岩屋港に到着。海岸沿いを走ってると、所々でバーベキューをやってる集団やら水上バイクを乗り回してる集団がいて、楽しそうやった。
移設された壁を、というか移設されるまで家の近くにあったのに、結局ちゃんと見なかったので、ちゃんと見たのは今回が初です。

そして、断層が保存されてるところのすぐ横のお家の庭。奥手のガラスの建物の中に断層が保存されてるんですが、写真撮影あかんと思って撮らんかったんです。きれいなまでに教科書どおりの断層やった。これもあれから15年くらいになりますが初めて見た(今まで見る機会がなかった)。手前のお家は、今は保存対象になって自由に中が見れるようになってるけど、地震後4年間住んではったということのようで。目の前はほんまに断層なんやけど、きれいに断層を避けて家が建ってたのがせめてもの救いなんやね。
地震体験ができるところは、揺れよりも音で個人的にダメですね。僕が居たところは震度4やったんで、揺れがどうのというよりも、あの朝早いときに地下から響き渡ってくる轟音で当時の出来事が思い出されてやるせない気持ちになる。
京都今村組のダンスをチラ見しながら、かえ氏お奨めという富島の焼きあなごを人かじりいただいてご飯が欲しくなった状態で、一同帰路へと就くことになりまして。

淡路島側から見た明石海峡大橋(アンカレイジパーク)。いつもは淡路島側からというと淡路SAになるんやけども、今日は下から。
料理部(現食物部)で見た、千代に行きたかった気持ちもなくはなかったけど、今度行きたい。みんなチャリで往復20kmも走って、筋肉痛にならんかなぁ。
投稿者 ただ : 22:28 | トラックバック (0) カテゴリー ; お出かけ
2009年5月 4日
 1時間では着かない
1時間では着かない
今日は金山組ボーリング…のはずやったけど、コミュニケーション不足により心斎橋に13時15分、と知らされたのが13時。15分で20kmを移動するのはどう考えても無理。おまけにTSUTAYAにレンタルしてたもんを返しとけと言付けされてるので、途中下車をしなあかんのは確実。
というわけでボーリング場に着いたのは14時半ごろ。無茶です。
後半戦の飲み会は、店の対応が残念やった。
投稿者 ただ : 23:11 | トラックバック (0) カテゴリー ; お出かけ
2009年5月 6日
 おまけの休日
おまけの休日
お墓へ行ってきた。
ついでに、ジャスコでクッションやら服やらを買い込んでおいた。車動かしてばっかりやけど、ほとんど行動範囲が変わらない。高速にも乗らないし。その代わり渋滞に巻き込まれることもないんですが。
GWが始まる前までこの日が振替休日やとは気付かなかったわけで。GW期間中はずっと家でPCも触らない代わりにRailsのプロジェクトを1つ考えてたりしてたけど、…それもだんだん飽きてきた。
そうそう、「Don't Say "Lazy"」といえばfetchをeargerにするかlazyにするかで判断が難しいんよね。Hibernateは2つ以上のカラムをEarger fetchできないから、もう、Earger fetchするのは諦めてるんやけど。Lazy fetchにしておいて、1+N問題はあるけどそこは耐えるしかない。
投稿者 ただ : 22:14 | トラックバック (0) カテゴリー ; mein Erbe
2009年5月 8日
 よくある失敗
よくある失敗
銀行に
行ったはものの
判留守番
…積み立ててる投信を特定口座にしようと思って、平日なのをいいことに銀行に勇んで行ったわけですが、一般口座のままだと何が不便なのかを確認することは結局聞かずに、住所変更せずに口座を切り替える方法はないのかということを聞いていた。住所変更は面倒なんですよ。今居るところに落ち着く気は全くないし、実家なんやから家が空というわけでもない。取りに行こうと思えば取りに行けるところに届く。お姉さんが言う「投資信託だから」というのにはどうにも理由がよく分からん。
で、住所変更を余儀なくされたわけだが判子を持って行ってない。まぁ判子を持たずに銀行に相談に行くこと自体失笑ものですが。
金融商品取引法でも、顧客に対して交付しなければならないことは書いてあっても、それがどこかということは触れてない。なんで指定したところに届けるんじゃあかんねやろう。顧客側が虚偽の申告をしたら金融機関側がまずいことになるんやろうか…そんなことはないと思うが。架空口座とかは問題かも知れんけど、それと投信口座は関係ないやん。それにちょっと調べれば本人の関係者が住んでるのは分かるわけやし。
登録住所と送付先住所を別指定できるとか、いまどきは電子交付があるんやから電子交付にするとか、もうちょっとええようにやってくれんもんやろうか。
投稿者 ただ : 22:21 | トラックバック (0) カテゴリー ; 投資
2009年5月10日
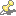 最近の外貨事情(-2009.04)
最近の外貨事情(-2009.04)
今年1月から4月までの損益状況とそのときの為替を重ねてみる。まぁ自分がどんな状況のときに利益出してるか(または大損してるか)が分かるわけですが。Self(赤)が自分の残高(指数化してて右軸で表示)、他は為替(こっちも指数化して左軸で表示)、どちらも初期値100で。

んー。なんやかんや言うても、結局はクロス円で動くなぁ。1月のオバマさん就任直後といい、4月後半の急激な円高といい…。しかも、EUR/*を使ってるもんだからきれいなまでにEUR/JPYに連動してる。
たけのこをまた掘った。前の倍以上を掘り出したもんで、右腕が大変なことに。
投稿者 ただ : 23:22 | トラックバック (0) カテゴリー ; 投資
2009年5月11日
 目の前にパトカーが
目の前にパトカーが
パトカーも
急ブレーキを
するんだな
昼ご飯を食べようと横断歩道で待ってたときのこと。
銀行に口座切り替えの用紙を送ったし、ひとまずこれで特定口座関連は無事終了。あ…もう1つ切り替えなあかんかった。
投稿者 ただ : 23:42 | トラックバック (0) カテゴリー ; mein Erbe
2009年5月13日
 Rails 2.3 on Mongrel + apache de prefix
Rails 2.3 on Mongrel + apache de prefix
prefixを付けてrailsプロジェクトを実行したいというのは、1つのサーバー内で複数のプロジェクトを構築するうえで必要になることなんですが、rails2.3になってから、Rackが使われるということもあり以前のような方法でprefixを使うことができなくなったんですよ…。
mongrelとapacheをつなげて使うときにはリバースプロキシを使ってやるのはRedmineを使えるようにするときに覚えたけど、Rails2.3でやるときには少々事情が異なってくる。というのも、prefixの扱いが変わったから。
Rails2.3になって、mongrel_cluster.ymlでのprefix指定をやめてActionController::Base.relative_url_rootに直接prefixを代入することになったけど、それをすると静的ファイルが読めなくなるという問題が起こるんよね…。Rackになってチンプンカンプンなことばっかり。WSGIとシステム的には同じになるとは分かってても…ねぇ。
Rack::Staticなんてあるからこれを使うもんやとばっかり思ってたから、全然解決せず困り果ててた。究極routes.rbにマッピングというのも…とは思ってやったものの、MimeTypeが原因でCSSがFirefoxでは適用できないという状態に。IEやとapplication/octet-streamでもちゃんと中身を見て解釈してくれるんですが。昨日あたりに、Rack::Staticのソースコードと実際のPATH_INFOを見てようやく解決の糸口が。
ProxyRequests Off
<Proxy http://ip.address/prefix/*>
Order deny,allow
Allow from all
</Proxy>#Rails 2.2まで ProxyPass /prefix http://localhost:3002/prefix
#Rails 2.2まで ProxyPassReverse /prefix http://localhost:3002/prefix
ProxyPass /prefix http://localhost:3002
ProxyPassReverse /prefix http://localhost:3002
ProxyPassかと。Rails2.2までのprefix指定やと"/prefix"が付いてないとHTMLにちゃんとURLを書いてくれなかったから(そのためにRedmineの画面が味気ないことになってしまった)、ないとあかんもんやと思ってたら、2.3では付けなくてよくなったんやね。これでRack::Staticなんか指定しなくてもちゃんと静的ファイルを取りにいってくれる。めでたしめでたし。
投稿者 ただ : 23:31 | トラックバック (0) カテゴリー ; プログラミングとか
2009年5月16日
 ついにやってきた
ついにやってきた
しかも、神戸に。でも…やっぱり高校生なのは何か特異性があるのかないのか。
初出のニュースでは神戸とはテロップで流れても神戸の「どこ」かが書いてないから"さっぱり意味がないニュースなんですが…"と思った。神戸のどこやねんと。感染者が出たことは事実として重要かも知れんけども。広いねんひとことで言われても…西から東に北まで(結局東やったけど)。
神戸に加えて茨木(+豊中)でも感染者が出たらしいので、生活圏とか考えたらもう1都市だけの問題じゃないやろうし。大阪湾沿岸と京都方面は危ないなぁ。…ここから動けない。
8日には既に感染してたということは、それより前の連休後半…東大阪の高校から感染者が出たときに成田で補足できなかった人が源?
兵庫高でも出たらしいので、これで完全に囲まれた。会社休みにならんかな。なったらなったで困るけど。今月末納期なんで…。
投稿者 ただ : 23:45 | トラックバック (0) カテゴリー ; mein Erbe
2009年5月19日
 マスクはどうしようか
マスクはどうしようか
大流行中っぽい状況ですが、自分の通勤ルートと時間を鑑みるに、マスクは要るのか? と。
ポートライナーなのでルート上に市民病院があるのは事実なんやけども、満員電車に乗ってるわけでもなく、ましてや駅を降りてからはほとんど人ごみとは縁のない状態。…どうしよう。
とりあえず、「抗ウィルスマスク」とかいう高級なマスクを昨日の昼に買ったんやけど(というかイズミヤにはこれしかなかった)、何がすごいのか、というと…目が細かいのね。「抗ウィルス」というからには抗体でも付いてるのかと思いきや、そんなんではないらしい。まぁ、抗原抗体反応が起こらないとウィルスの動きを封じられないのは分かってるが。マスクに抗体ばら撒いたのが作れることは作れるってテレビで見たけど、そういうのがこの冬流行ったりして。問題は何に対する抗体をばら撒くか…か。
ウィルス不活化するとかいう空気清浄機が出回ってるくらいやから、高級マスクと同じくらいの値段(@330)で作れるんじゃね?
で、マスクは要らんのとちゃうか…というのが僕自身の感想。阪急とかJRとかに乗るときはつけるやろうけど。ポートライナーは夜10時半を回ったくらいのに乗ればマスクは要らん。イズミヤも今週は「不要不急の外出は控えて」る人が多くなった影響で比較的空いてるし。というかマスクは苦手です。
投稿者 ただ : 21:28 | トラックバック (0) カテゴリー ; mein Erbe
2009年5月20日
 EMBOSSを試す その2
EMBOSSを試す その2 
さて、インストールしたEMBOSS、試しに何か使ってみようといいネタ探していたらseqretというコマンドがよさそうなのでこれを試す。seqretは、配列をFASTA形式で取得するコマンドのよう。
$ seqret --help
Standard (Mandatory) qualifiers:
[-sequence] seqall (Gapped) sequence(s) filename and optional
format, or reference (input USA)
[-outseq] seqoutall [. ] Sequence set(s)
filename and optional format (output USA)Additional (Optional) qualifiers: (none)
Advanced (Unprompted) qualifiers:
-feature boolean Use feature information
-firstonly boolean [N] Read one sequence and stopGeneral qualifiers:
-help boolean Report command line options. More
information on associated and general
qualifiers can be found with -help -verbose
ちょうど、巷で話題になっている豚インフルの配列が公開されているので、それを取ってみることに。
$ seqret genbank:GQ149630
Reads and writes (returns) sequences
output sequence(s) [gq149630.fasta]:
このプログラム、ファイルとして出力されるようにプログラムされていて標準出力には出てこない。
$ cat gq149630.fasta
>GQ149630 GQ149630.1 Influenza A virus (A/Mexico/4603/2009(H1N1)) segment 4 hemagglutinin (HA) gene, complete cds.
atgaaggcaatactagtagttctgctatatacatttgcaaccgcaaatgcagacacatta
tgtataggttatcatgcgaacaattcaacagacactgtagacacagtactagaaaagaat
gtaacagtaacacactctgttaaccttctagaagacaagcataacgggaaactatgcaaa
ctaagaggggtagccccattgcatttgggtaaatgtaacattgctggctggatcctggga
aatccagagtgtgaatcactctccacagcaagctcatggtcctacattgtggaaacatct
agttcagacaatggaacgtgttacccaggagatttcatcgattatgaggagctaagagag
caattgagctcagtgtcatcatttgaaaggtttgagatattccccaagacaagttcatgg
cccaatcatgactcgaacaaaggtgtaacggcagcatgtcctcatgctggagcaaaaagc
ttctacaaaaatttaatatggctagttaaaaaaggaaattcatacccaaagctcagcaaa
tcctacattaatgataaagggaaagaagtcctcgtgctatggggcattcaccatccatct
actagtgctgaccaacaaagtctctatcagaatgcagatgcatatgtttttgtggggtca
tcaagatacagcaagaagttcaagccggaaatagcaataagacccaaagtgagggatcaa
gaagggagaatgaactattactggacactagtagagccgggagacaaaataacattcgaa
gcaactggaaatctagtggtaccgagatatgcattcgcaatggaaagaaatgctggatct
ggtattatcatttcagatacaccagtccacgattgcaatacaacttgtcagacacccaag
ggtgctataaacaccagcctcccatttcagaatatacatccgatcacaattggaaaatgt
ccaaaatatgtaaaaagcacaaaattgagactggccacaggattgaggaatgtcccgtct
attcaatctagaggcctatttggggccattgccggtttcattgaaggggggtggacaggg
atggtagatggatggtacggttatcaccatcaaaatgagcaggggtcaggatatgcagcc
gacctgaagagcacacagaatgccattgacgagattactaacaaagtaaattctgttatt
gaaaagatgaatacacagttcacagcagtaggtaaagagttcaaccacctggaaaaaaga
atagagaatttaaataaaaaagttgatgatggtttcctggacatttggacttacaatgcc
gaactgttggttctattggaaaatgaaagaactttggactaccacgattcaaatgtgaag
aacttatatgaaaaggtaagaagccagttaaaaaacaatgccaaggaaattggaaacggc
tgctttgaattttaccacaaatgcgataacacgtgcatggaaagtgtcaaaaatgggact
tatgactacccaaaatactcagaggaagcaaaattaaacagagaagaaatagatggggta
aagctggaatcaacaaggatttaccagattttggcgatctattcaactgtcgccagttca
ttggtactggtagtctccctgggggcaatcagtttctggatgtgctctaatgggtctcta
cagtgtagaatatgtatttaa
かと思ったら標準出力にも出せた($seqret genbank:GQ149630 stdout)。entretを使うと、全部のデータを出せるらしい。
$ entret genbank:GQ149630 stdout
Retrieves sequence entries from flatfile databases and files
LOCUS GQ149630 1701 bp cRNA linear VRL 13-MAY-2009
DEFINITION Influenza A virus (A/Mexico/4603/2009(H1N1)) segment 4
hemagglutinin (HA) gene, complete cds.
ACCESSION GQ149630
VERSION GQ149630.1 GI:237511793
DBLINK Project:37813
KEYWORDS .
SOURCE Influenza A virus (A/Mexico/4603/2009(H1N1))
ORGANISM Influenza A virus (A/Mexico/4603/2009(H1N1))
Viruses; ssRNA negative-strand viruses; Orthomyxoviridae;
Influenzavirus A.
REFERENCE 1 (bases 1 to 1701)
AUTHORS Alpuche-Aranda,C.M., Lopez-Gatell,H., Olivera,H., Lopez,I., Shu,B.,
Balish,A., Garten,R., Smith,C., Emery,S., Barnes,J., Deyde,V.,
Klimov,A. and Cox,N.
TITLE Human infection with novel swine H1N1 influenza
JOURNAL Unpublished
REFERENCE 2 (bases 1 to 1701)
AUTHORS Alpuche-Aranda,C.M., Lopez-Gatell,H., Olivera,H., Lopez,I., Shu,B.,
Balish,A., Garten,R., Smith,C., Emery,S., Barnes,J., Deyde,V.,
Klimov,A. and Cox,N.
TITLE Direct Submission
JOURNAL Submitted (13-MAY-2009) WHO Collaborating Center for Surveillance,
Epidemiology and Control of Influenza, Influenza Division, Centers
for Disease Control and Prevention, 1600 Clifton Road NE, Atlanta,
GA 30333, USA
COMMENT Swine influenza A (H1N1) virus isolated during human swine flu
outbreak of 2009.Some of the information does not have GenBank feature identifiers
and is being provided in the comment section.##EpifluData-START##
Isolate A/Mexico/4603/2009
Subtype H1N1
Segment_name HA
Host_gender F
Host_age 59
Passage_history C1
Adamantane_resistance resistant
Zanamivir_resistance sensitive
Oseltamivir_resistance sensitive
Country Mexico
Collection_day 19
Collection_month 4
Collection_year 2009
EPI_accession EPI176604
##EpifluData-END##
FEATURES Location/Qualifiers
source 1..1701
/organism="Influenza A virus (A/Mexico/4603/2009(H1N1))"
/mol_type="viral cRNA"
/strain="A/Mexico/4603/2009"
/serotype="H1N1"
/host="Homo sapiens; gender F; age 59"
/db_xref="taxon:644887"
/segment="4"
/country="Mexico"
/collection_date="19-Apr-2009"
gene 1..1701
/gene="HA"
CDS 1..1701
/gene="HA"
/codon_start=1
/product="hemagglutinin"
/protein_id="ACQ99608.1"
/db_xref="GI:237511794"
/translation="MKAILVVLLYTFATANADTLCIGYHANNSTDTVDTVLEKNVTVT
HSVNLLEDKHNGKLCKLRGVAPLHLGKCNIAGWILGNPECESLSTASSWSYIVETSSS
DNGTCYPGDFIDYEELREQLSSVSSFERFEIFPKTSSWPNHDSNKGVTAACPHAGAKS
FYKNLIWLVKKGNSYPKLSKSYINDKGKEVLVLWGIHHPSTSADQQSLYQNADAYVFV
GSSRYSKKFKPEIAIRPKVRDQEGRMNYYWTLVEPGDKITFEATGNLVVPRYAFAMER
NAGSGIIISDTPVHDCNTTCQTPKGAINTSLPFQNIHPITIGKCPKYVKSTKLRLATG
LRNVPSIQSRGLFGAIAGFIEGGWTGMVDGWYGYHHQNEQGSGYAADLKSTQNAIDEI
TNKVNSVIEKMNTQFTAVGKEFNHLEKRIENLNKKVDDGFLDIWTYNAELLVLLENER
TLDYHDSNVKNLYEKVRSQLKNNAKEIGNGCFEFYHKCDNTCMESVKNGTYDYPKYSE
EAKLNREEIDGVKLESTRIYQILAIYSTVASSLVLVVSLGAISFWMCSNGSLQCRICI"
ORIGIN
1 atgaaggcaa tactagtagt tctgctatat acatttgcaa ccgcaaatgc agacacatta
61 tgtataggtt atcatgcgaa caattcaaca gacactgtag acacagtact agaaaagaat
121 gtaacagtaa cacactctgt taaccttcta gaagacaagc ataacgggaa actatgcaaa
181 ctaagagggg tagccccatt gcatttgggt aaatgtaaca ttgctggctg gatcctggga
241 aatccagagt gtgaatcact ctccacagca agctcatggt cctacattgt ggaaacatct
301 agttcagaca atggaacgtg ttacccagga gatttcatcg attatgagga gctaagagag
361 caattgagct cagtgtcatc atttgaaagg tttgagatat tccccaagac aagttcatgg
421 cccaatcatg actcgaacaa aggtgtaacg gcagcatgtc ctcatgctgg agcaaaaagc
481 ttctacaaaa atttaatatg gctagttaaa aaaggaaatt catacccaaa gctcagcaaa
541 tcctacatta atgataaagg gaaagaagtc ctcgtgctat ggggcattca ccatccatct
601 actagtgctg accaacaaag tctctatcag aatgcagatg catatgtttt tgtggggtca
661 tcaagataca gcaagaagtt caagccggaa atagcaataa gacccaaagt gagggatcaa
721 gaagggagaa tgaactatta ctggacacta gtagagccgg gagacaaaat aacattcgaa
781 gcaactggaa atctagtggt accgagatat gcattcgcaa tggaaagaaa tgctggatct
841 ggtattatca tttcagatac accagtccac gattgcaata caacttgtca gacacccaag
901 ggtgctataa acaccagcct cccatttcag aatatacatc cgatcacaat tggaaaatgt
961 ccaaaatatg taaaaagcac aaaattgaga ctggccacag gattgaggaa tgtcccgtct
1021 attcaatcta gaggcctatt tggggccatt gccggtttca ttgaaggggg gtggacaggg
1081 atggtagatg gatggtacgg ttatcaccat caaaatgagc aggggtcagg atatgcagcc
1141 gacctgaaga gcacacagaa tgccattgac gagattacta acaaagtaaa ttctgttatt
1201 gaaaagatga atacacagtt cacagcagta ggtaaagagt tcaaccacct ggaaaaaaga
1261 atagagaatt taaataaaaa agttgatgat ggtttcctgg acatttggac ttacaatgcc
1321 gaactgttgg ttctattgga aaatgaaaga actttggact accacgattc aaatgtgaag
1381 aacttatatg aaaaggtaag aagccagtta aaaaacaatg ccaaggaaat tggaaacggc
1441 tgctttgaat tttaccacaa atgcgataac acgtgcatgg aaagtgtcaa aaatgggact
1501 tatgactacc caaaatactc agaggaagca aaattaaaca gagaagaaat agatggggta
1561 aagctggaat caacaaggat ttaccagatt ttggcgatct attcaactgt cgccagttca
1621 ttggtactgg tagtctccct gggggcaatc agtttctgga tgtgctctaa tgggtctcta
1681 cagtgtagaa tatgtattta a
//
投稿者 ただ : 23:00 | トラックバック (4) カテゴリー ; 生命科学
2009年5月21日
 EMBOSSを試す その3
EMBOSSを試す その3
wossnameコマンドを用いると、利用可能なコマンドと簡単な説明が一覧で出てくるらしい。とりあえず出してみた。
$ wossname
Finds programs by keywords in their short description
Text to search for, or blank to list all programs:
ACD
acdc Test an application ACD file
acdpretty Correctly reformat an application ACD file
acdtable Generate an HTML table of parameters from an application ACD file
acdtrace Trace processing of an application ACD file (for testing)
acdvalid Validate an application ACD fileALIGNMENT CONSENSUS
cons Create a consensus sequence from a multiple alignment
consambig Create an ambiguous consensus sequence from a multiple alignment
megamerger Merge two large overlapping DNA sequences
merger Merge two overlapping sequencesALIGNMENT DIFFERENCES
diffseq Compare and report features of two similar sequencesALIGNMENT DOT PLOTS
dotmatcher Draw a threshold dotplot of two sequences
dotpath Draw a non-overlapping wordmatch dotplot of two sequences
dottup Displays a wordmatch dotplot of two sequences
polydot Draw dotplots for all-against-all comparison of a sequence setALIGNMENT GLOBAL
est2genome Align EST sequences to genomic DNA sequence
needle Needleman-Wunsch global alignment of two sequences
stretcher Needleman-Wunsch rapid global alignment of two sequencesALIGNMENT LOCAL
matcher Waterman-Eggert local alignment of two sequences
seqmatchall All-against-all word comparison of a sequence set
supermatcher Calculate approximate local pair-wise alignments of larger sequences
water Smith-Waterman local alignment of sequences
wordfinder Match large sequences against one or more other sequences
wordmatch Finds regions of identity (exact matches) of two sequencesALIGNMENT MULTIPLE
edialign Local multiple alignment of sequences
emma Multiple sequence alignment (ClustalW wrapper)
infoalign Display basic information about a multiple sequence alignment
plotcon Plot conservation of a sequence alignment
prettyplot Draw a sequence alignment with pretty formatting
showalign Display a multiple sequence alignment in pretty format
tranalign Generate an aligment of nucleic coding regions from aligned proteinsDISPLAY
abiview Display the trace in an ABI sequencer file
cirdna Draws circular maps of DNA constructs
lindna Draws linear maps of DNA constructs
pepnet Draw a helical net for a protein sequence
pepwheel Draw a helical wheel diagram for a protein sequence
prettyplot Draw a sequence alignment with pretty formatting
prettyseq Write a nucleotide sequence and its translation to file
remap Display restriction enzyme binding sites in a nucleotide sequence
seealso Finds programs with similar function to a specified program
showalign Display a multiple sequence alignment in pretty format
showdb Displays information on configured databases
showfeat Display features of a sequence in pretty format
showseq Displays sequences with features in pretty format
sixpack Display a DNA sequence with 6-frame translation and ORFs
textsearch Search the textual description of sequence(s)EDIT
aligncopy Reads and writes alignments
aligncopypair Reads and writes pairs from alignments
biosed Replace or delete sequence sections
codcopy Copy and reformat a codon usage table
cutseq Removes a section from a sequence
degapseq Removes non-alphabetic (e.g. gap) characters from sequences
descseq Alter the name or description of a sequence
entret Retrieves sequence entries from flatfile databases and files
extractalign Extract regions from a sequence alignment
extractfeat Extract features from sequence(s)
extractseq Extract regions from a sequence
featcopy Reads and writes a feature table
featreport Reads and writes a feature table
listor Write a list file of the logical OR of two sets of sequences
makenucseq Create random nucleotide sequences
makeprotseq Create random protein sequences
maskambignuc Masks all ambiguity characters in nucleotide sequences with N
maskambigprot Masks all ambiguity characters in protein sequences with X
maskfeat Write a sequence with masked features
maskseq Write a sequence with masked regions
newseq Create a sequence file from a typed-in sequence
nohtml Remove mark-up (e.g. HTML tags) from an ASCII text file
noreturn Remove carriage return from ASCII files
nospace Remove all whitespace from an ASCII text file
notab Replace tabs with spaces in an ASCII text file
notseq Write to file a subset of an input stream of sequences
nthseq Write to file a single sequence from an input stream of sequences
pasteseq Insert one sequence into another
revseq Reverse and complement a nucleotide sequence
seqret Reads and writes (returns) sequences
seqretsplit Reads sequences and writes them to individual files
sizeseq Sort sequences by size
skipredundant Remove redundant sequences from an input set
skipseq Reads and writes (returns) sequences, skipping first few
splitter Split sequence(s) into smaller sequences
trimest Remove poly-A tails from nucleotide sequences
trimseq Remove unwanted characters from start and end of sequence(s)
union Concatenate multiple sequences into a single sequence
vectorstrip Removes vectors from the ends of nucleotide sequence(s)
yank Add a sequence reference (a full USA) to a list fileENZYME KINETICS
findkm Calculate and plot enzyme reaction dataFEATURE TABLES
coderet Extract CDS, mRNA and translations from feature tables
extractfeat Extract features from sequence(s)
maskfeat Write a sequence with masked features
showfeat Display features of a sequence in pretty format
twofeat Finds neighbouring pairs of features in sequence(s)INFORMATION
infoalign Display basic information about a multiple sequence alignment
infoseq Display basic information about sequences
seealso Finds programs with similar function to a specified program
showdb Displays information on configured databases
textsearch Search the textual description of sequence(s)
tfm Displays full documentation for an application
whichdb Search all sequence databases for an entry and retrieve it
wossname Finds programs by keywords in their short descriptionNUCLEIC 2D STRUCTURE
einverted Finds inverted repeats in nucleotide sequencesNUCLEIC CODON USAGE
cai Calculate codon adaptation index
chips Calculates Nc codon usage statistic
codcmp Codon usage table comparison
cusp Create a codon usage table from nucleotide sequence(s)
syco Draw synonymous codon usage statictic plot for a nucleotide sequenceNUCLEIC COMPOSITION
banana Plot bending and curvature data for B-DNA
btwisted Calculate the twisting in a B-DNA sequence
chaos Draw a chaos game representation plot for a nucleotide sequence
compseq Calculate the composition of unique words in sequences
dan Calculates nucleic acid melting temperature
density Draw a nucleic acid density plot
freak Generate residue/base frequency table or plot
isochore Plots isochores in DNA sequences
sirna Finds siRNA duplexes in mRNA
wordcount Count and extract unique words in DNA sequence(s)NUCLEIC CPG ISLANDS
cpgplot Identify and plot CpG islands in nucleotide sequence(s)
cpgreport Identify and report CpG-rich regions in nucleotide sequence(s)
geecee Calculate fractional GC content of nucleic acid sequences
newcpgreport Identify CpG islands in nucleotide sequence(s)
newcpgseek Identify and report CpG-rich regions in nucleotide sequence(s)NUCLEIC GENE FINDING
getorf Finds and extracts open reading frames (ORFs)
marscan Finds matrix/scaffold recognition (MRS) signatures in DNA sequences
plotorf Plot potential open reading frames in a nucleotide sequence
showorf Display a nucleotide sequence and translation in pretty format
sixpack Display a DNA sequence with 6-frame translation and ORFs
syco Draw synonymous codon usage statictic plot for a nucleotide sequence
tcode Identify protein-coding regions using Fickett TESTCODE statistic
wobble Plot third base position variability in a nucleotide sequenceNUCLEIC MOTIFS
dreg Regular expression search of nucleotide sequence(s)
fuzznuc Search for patterns in nucleotide sequences
fuzztran Search for patterns in protein sequences (translated)
marscan Finds matrix/scaffold recognition (MRS) signatures in DNA sequencesNUCLEIC MUTATION
msbar Mutate a sequence
shuffleseq Shuffles a set of sequences maintaining compositionNUCLEIC PRIMERS
eprimer3 Picks PCR primers and hybridization oligos
primersearch Search DNA sequences for matches with primer pairs
stssearch Search a DNA database for matches with a set of STS primersNUCLEIC PROFILES
profit Scan one or more sequences with a simple frequency matrix
prophecy Create frequency matrix or profile from a multiple alignment
prophet Scan one or more sequences with a Gribskov or Henikoff profileNUCLEIC REPEATS
einverted Finds inverted repeats in nucleotide sequences
equicktandem Finds tandem repeats in nucleotide sequences
etandem Finds tandem repeats in a nucleotide sequence
palindrome Finds inverted repeats in nucleotide sequence(s)NUCLEIC RESTRICTION
recoder Find restriction sites to remove (mutate) with no translation change
redata Retrieve information from REBASE restriction enzyme database
remap Display restriction enzyme binding sites in a nucleotide sequence
restover Find restriction enzymes producing a specific overhang
restrict Report restriction enzyme cleavage sites in a nucleotide sequence
showseq Displays sequences with features in pretty format
silent Find restriction sites to insert (mutate) with no translation changeNUCLEIC TRANSCRIPTION
jaspscan Scans DNA sequences for transcription factors
tfscan Identify transcription factor binding sites in DNA sequencesNUCLEIC TRANSLATION
backtranambig Back-translate a protein sequence to ambiguous nucleotide sequence
backtranseq Back-translate a protein sequence to a nucleotide sequence
coderet Extract CDS, mRNA and translations from feature tables
plotorf Plot potential open reading frames in a nucleotide sequence
prettyseq Write a nucleotide sequence and its translation to file
remap Display restriction enzyme binding sites in a nucleotide sequence
showorf Display a nucleotide sequence and translation in pretty format
showseq Displays sequences with features in pretty format
sixpack Display a DNA sequence with 6-frame translation and ORFs
transeq Translate nucleic acid sequencesPHYLOGENY MOLECULAR SEQUENCE
distmat Create a distance matrix from a multiple sequence alignmentPROTEIN 2D STRUCTURE
garnier Predicts protein secondary structure using GOR method
helixturnhelix Identify nucleic acid-binding motifs in protein sequences
hmoment Calculate and plot hydrophobic moment for protein sequence(s)
pepcoil Predicts coiled coil regions in protein sequences
pepnet Draw a helical net for a protein sequence
pepwheel Draw a helical wheel diagram for a protein sequence
tmap Predict and plot transmembrane segments in protein sequencesPROTEIN 3D STRUCTURE
psiphi Calculates phi and psi torsion angles from protein coordinatesPROTEIN COMPOSITION
backtranambig Back-translate a protein sequence to ambiguous nucleotide sequence
backtranseq Back-translate a protein sequence to a nucleotide sequence
charge Draw a protein charge plot
checktrans Reports STOP codons and ORF statistics of a protein
compseq Calculate the composition of unique words in sequences
emowse Search protein sequences by digest fragment molecular weight
freak Generate residue/base frequency table or plot
iep Calculate the isoelectric point of proteins
mwcontam Find weights common to multiple molecular weights files
mwfilter Filter noisy data from molecular weights file
octanol Draw a White-Wimley protein hydropathy plot
pepinfo Plot amino acid properties of a protein sequence in parallel
pepstats Calculates statistics of protein properties
pepwindow Draw a Kyte-Doolittle hydropathy plot for a protein sequence
pepwindowall Draw Kyte-Doolittle hydropathy plot for a protein alignment
wordcount Count and extract unique words in DNA sequence(s)PROTEIN MOTIFS
antigenic Finds antigenic sites in proteins
digest Reports on protein proteolytic enzyme or reagent cleavage sites
epestfind Finds PEST motifs as potential proteolytic cleavage sites
fuzzpro Search for patterns in protein sequences
fuzztran Search for patterns in protein sequences (translated)
helixturnhelix Identify nucleic acid-binding motifs in protein sequences
oddcomp Identify proteins with specified sequence word composition
patmatdb Searches protein sequences with a sequence motif
patmatmotifs Scan a protein sequence with motifs from the PROSITE database
pepcoil Predicts coiled coil regions in protein sequences
preg Regular expression search of protein sequence(s)
pscan Scans protein sequence(s) with fingerprints from the PRINTS database
sigcleave Reports on signal cleavage sites in a protein sequencePROTEIN MUTATION
msbar Mutate a sequence
shuffleseq Shuffles a set of sequences maintaining compositionPROTEIN PROFILES
profit Scan one or more sequences with a simple frequency matrix
prophecy Create frequency matrix or profile from a multiple alignment
prophet Scan one or more sequences with a Gribskov or Henikoff profileUTILS DATABASE CREATION
aaindexextract Extract amino acid property data from AAINDEX
cutgextract Extract codon usage tables from from CUTG database
jaspextract Extract data from JASPAR
printsextract Extract data from PRINTS database for use by pscan
prosextract Processes the PROSITE motif database for use by patmatmotifs
rebaseextract Process the REBASE database for use by restriction enzyme applications
tfextract Process TRANSFAC transcription factor database for use by tfscanUTILS DATABASE INDEXING
dbiblast Index a BLAST database
dbifasta Index a fasta file database
dbiflat Index a flat file database
dbigcg Index a GCG formatted database
dbxfasta Index a fasta file database using b+tree indices
dbxflat Index a flat file database using b+tree indices
dbxgcg Index a GCG formatted database using b+tree indicesUTILS MISC
embossdata Find and retrieve EMBOSS data files
embossversion Writes the current EMBOSS version number to a file
投稿者 ただ : 01:11 | トラックバック (0) カテゴリー ; 生命科学
2009年5月22日
 EMBOSSを試す その4
EMBOSSを試す その4 
今度は、アラインメント(ref. Wikipedia)してみます。stretcherコマンドは、Needleman-Wunsch法を使った大域的アライメントの実装です。NCBIの相同遺伝子配列の検索で有名なBLASTは配列の相同性をスコアリングするのに局所的アライメントのSmith-Waterman法を使っています。
以前参考にした、genbank:GQ149630(Influenza A virus (A/Mexico/4603/2009(H1N1)) segment 4 hemagglutinin (HA) gene, complete cds.)と、genbank:GQ169382(Influenza A virus (A/Thailand/104/2009(H1N1)) segment 4 hemagglutinin (HA) gene, complete cds.)を比べてみます。どちらも、豚インフルのHAタンパクをコードしている遺伝子で、違いはメキシコで採れたものかタイで採れたものか、です。
$ stretcher genbank:GQ169382 genbank:GQ149630
Needleman-Wunsch rapid global alignment of two sequences
Output alignment [gq169382.stretcher]: stdout
########################################
# Program: stretcher
# Rundate: Sun 24 May 2009 00:23:40
# Commandline: stretcher
# [-asequence] genbank:GQ169382
# [-bsequence] genbank:GQ149630
# -outfile stdout
# Align_format: markx0
# Report_file: stdout
#########################################=======================================
#
# Aligned_sequences: 2
# 1: GQ169382
# 2: GQ149630
# Matrix: EDNAFULL
# Gap_penalty: 16
# Extend_penalty: 4
#
# Length: 1741
# Identity: 1699/1741 (97.6%)
# Similarity: 1699/1741 (97.6%)
# Gaps: 40/1741 ( 2.3%)
# Score: 8303
#
#
#=======================================10 20 30 40 50
GQ1693 GCAAAAGCAGGGGAAAACAAAAGCACAAAATGAAGGCAATACTAGTAGTT
:::::::::::::::::::::
GQ1496 -----------------------------ATGAAGGCAATACTAGTAGTT
10 20..(略)..
1660 1670 1680 1690 1700
GQ1693 TGGTACTGGTAGTCTCCCTGGGGGCAATCAGTTTCTGGATGTGCTCTAAT
::::::::::::::::::::::::::::::::::::::::::::::::::
GQ1496 TGGTACTGGTAGTCTCCCTGGGGGCAATCAGTTTCTGGATGTGCTCTAAT
1630 1640 1650 1660 16701710 1720 1730 1740
GQ1693 GGGTCTCTACAGTGTAGAATATGTATTTAACCATAGGATTC
::::::::::::::::::::::::::::::
GQ1496 GGGTCTCTACAGTGTAGAATATGTATTTAA-----------
1680 1690 1700
#---------------------------------------
#---------------------------------------
ほとんど一緒、というか前後に余分なところがあるだけで重なるところは全部同じですね。ある意味当然の結果なんですが(でないと遺伝子検査とかできないしね)。HAタンパクではない、別のタンパクをコードしている遺伝子と比較します。genbank:GQ169382と比較する、genbank:GQ169385(Influenza A virus (A/Thailand/104/2009(H1N1)) segment 5 nucleocapsid protein (NP) gene, complete cds.)は、採った検体は同じで、コードしているタンパクが違います。見た感じが明らかに違って、スコアも低いです。
$ stretcher genbank:GQ169382 genbank:GQ169385
Needleman-Wunsch rapid global alignment of two sequences
Output alignment [gq169382.stretcher]: stdout
########################################
# Program: stretcher
# Rundate: Sun 24 May 2009 00:48:51
# Commandline: stretcher
# [-asequence] genbank:GQ169382
# [-bsequence] genbank:GQ169385
# -outfile stdout
# Align_format: markx0
# Report_file: stdout
#########################################=======================================
#
# Aligned_sequences: 2
# 1: GQ169382
# 2: GQ169385
# Matrix: EDNAFULL
# Gap_penalty: 16
# Extend_penalty: 4
#
# Length: 1779
# Identity: 840/1779 (47.2%)
# Similarity: 840/1779 (47.2%)
# Gaps: 260/1779 (14.6%)
# Score: -732
#
#
#=======================================10 20 30 40 50
GQ1693 GCAAAAGCAGGGGAAAACAAAAGCACAAAATGAAGGCAATACTAGTAGTT
:::: : : :: ::: : ::: : :: ::
GQ1693 --------AGGGTAGA---TAATCACTCACTGAGTGACATCGAAG-----
10 20 3060 70 80 90 100
GQ1693 CTGCTATATACATTTGCAACCGCAAATGCAGACACATTATGTATAGGTTA
: : :: : :: ::: : : :
GQ1693 ----------------CCATGGCGTCTCAAGGCACCAAACG--------A
40 50 60..(中略)..
1670 1680 1690 1700 1710
GQ1693 TCCCTGGGGGCAATCAGTTTCT--GGATGTGCTCTAATGGGTCTCTACAG
:: :: : : ::::: ::: : : :: ::::
GQ1693 --AATGAAGGGTCTTA-TTTCTTCGGAGACAATGCAGAGGAGTATGACAG
1490 1500 1510 15201720 1730 1740
GQ1693 TGTAGAATATGTATTTAACCATAGGATTC
: :: : : :: : : :
GQ1693 TTGAGGAAAAATACCCTTGTTTCT-ACTA
1530 1540 1550
#---------------------------------------
#---------------------------------------
これだけでは何のことやら、かもしれませんが。新型インフルエンザA(H1N1)の流行状況-更新4(ref. 国立感染症研究所 感染症情報センター)を見ると、豚インフルの遺伝子の特徴を調べるのに使っているのが分かるでしょう。もっとも、こんなのは総当りで調べてたら見る方もしんどいのでまとめて調べてるんでしょうけど。
ウイルス学的状況
今回の新型インフルエンザ(Swine-origin influenza A/H1N1)のウイルス学的な解析においては、本ウイルスは、1930年代以降に発見された米国由来のブタインフルエンザウイルス、ヒトインフルエンザウイルス(H3N2)、トリインフルエンザウイルスの3つのウイルスの内部遺伝子が遺伝子再集合をおこしたTriple Reassortantに、更にアジア-ユーラシア由来のブタインフルエンザウイルスの遺伝子分節が含まれている。この遺伝子がどのようにメキシコまで到達したかは不明である。現在のすべての遺伝子分節はブタ型の特徴を表しており、ヒト型への適応はみられていないとされている。3月以降に分離された30株ほどのウイルス遺伝子を調べた結果、99%の遺伝子が同一であったことから、変異の速度が速いRNAウイルスとしての性質を考えた場合、最近発生したウイルスであると推定されている。これまでに複数の国の患者から分離されたウイルスは現時点では非常に類似していると考えられ、引き続きノイラミニダーゼ阻害剤に対する感受性を維持している。
投稿者 ただ : 23:15 | トラックバック (1) カテゴリー ; 生命科学
2009年5月24日
 EMBOSSを試す その5
EMBOSSを試す その5
前回のその4で、1対1の配列を比較するアラインメントをやりました。こんどはまとめてやるマルチプルアラインメントです。これをやるのはいろんなプログラムがあるんですが、ClustalWのラッパーというemmaではなく、edialignというコマンドでやります。
- FJ981612 FJ981612.1 Influenza A virus (A/Texas/04/2009(H1N1)) segment 4 hemagglutinin (HA) gene, complete cds.
- GQ117112 GQ117112.1 Influenza A virus (A/Michigan/02/2009(H1N1)) segment 4 hemagglutinin (HA) gene, complete cds.
- GQ131023 GQ131023.1 Influenza A virus (A/Korea/01/2009(H1N1)) segment 4 hemagglutinin (HA) gene, complete cds.
- AB013810 AB013810.1 Influenza A virus (A/Tokyo/1567/98(H3N2)) gene for hemagglutinin, partial cds.
- AB043499 AB043499.1 Influenza A virus (A/Yokohama/24/2000(H1N1)) HA gene for hemagglutinin, partial cds.
- AF144305 AF144305.1 Influenza A virus (A/Goose/Guangdong/1/96(H5N1)) hemagglutinin (HA) gene, complete cds.
今回は上の配列を用意しておきます。上から順に3つは豚インフル(H1N1)、香港型(H3N2)、ソ連型(H1N1)、鳥インフル(H5N1)です。どれもHAタンパクの遺伝子です。H1, H3, H5というのは、このHAタンパクの種類で分かれてることになってます。これを局所的アライメントで相同性を比べてみます。
DIALIGN 2.2.1
*************Program code written by Burkhard Morgenstern and Said Abdeddaim
e-mail contact: dialign (at) gobics (dot) dePublished research assisted by DIALIGN 2 should cite:
Burkhard Morgenstern (1999).
DIALIGN 2: improvement of the segment-to-segment
approach to multiple sequence alignment.
Bioinformatics 15, 211 - 218.For more information, please visit the DIALIGN home page at
http://bibiserv.techfak.uni-bielefeld.de/dialign/
************************************************************
DIALIGNというプログラムが使われているようです。
program call: edialign in.seq stdout
Aligned sequences: length:
================== =======1) FJ981612 1701
2) GQ117112 1701
3) GQ131023 1701
4) AB013810 710
5) AB043499 1032
6) AF144305 1760Average seq. length: 1434.2
Please note that only upper-case letters are considered to be aligned.
Alignment (DIALIGN format):
===========================FJ981612 1 ---------- ---------- ---------- ---------- ----------
GQ117112 1 ---------- ---------- ---------- ---------- ----------
GQ131023 1 ---------- ---------- ---------- ---------- ----------
AB013810 1 ttgttgaacg cagcaaagct tacagcaact gttaccctta tgatgtgccg
AB043499 1 ---------- ---------- ---------- ---------- ----------
AF144305 1 gca------- ---------- ---------- ---------- ----------0000000000 0000000000 0000000000 0000000000 0000000000
FJ981612 1 ---------- ---------- ---------- ---------- ----------
GQ117112 1 ---------- ---------- ---------- ---------- ----------
GQ131023 1 ---------- ---------- ---------- ---------- ----------
AB013810 51 gattatgcct cccttaggtc actagttgcc tcatccggca ccctggagtt
AB043499 1 ---------- ---------- ---------- ---------- ----------
AF144305 4 ---------- ---------- ---------- ---------- ----------0000000000 0000000000 0000000000 0000000000 0000000000
FJ981612 1 -----ATGAA GGCAATACTA GTAGTTCTGC TATATAC--- ----------
GQ117112 1 -----ATGAA GGCAATACTA GTAGTTCTGC TATATAC--- ----------
GQ131023 1 -----ATGAA GGCAATACTA GTAGTTCTGC TATATAC--- ----------
AB013810 101 taacaATGAA AGCttcaatt GGACTGGAGT CGCTCAGAAT GGAACAAGCT
AB043499 1 -----ATGAA AGCAAAACTA CTAGTTCTGT TGTGTGC--- ----------
AF144305 4 ---------- ---------- GGGGTATAAT CTGTCAAAAT GGAGAAAATA0000055555 5555555555 5555555555 5555555000 0000000000
FJ981612 33 ---------- ---------- ATTTGCAACC GCAAATGCAG ACACATTATG
GQ117112 33 ---------- ---------- ATTTGCAACC GCAAATGCAG ACACATTATG
GQ131023 33 ---------- ---------- ATTTGCAACC GCAAATGCAG ACACATTATG
AB013810 151 TTGCTTgcaa aaggagatct ATTAAAAGTT TCTTTAGTAG ATTGAATTGG
AB043499 33 ---------- ---------- ATTTACAGCT ACATATGCAG ACACAATATG
AF144305 34 GTGCTTcttc ttgcaatagt cagtcttgtc aaaagtgatc ag---ATTTG0000000000 0000000000 5555555444 4777777777 7777777788
FJ981612 63 TATAGGTTAT CATGCGAACA ATTCAACAGA CACTGTAGAC ACAGTACTAG
GQ117112 63 TATAGGTTAT CATGCGAACA ATTCAACAGA CACTGTAGAC ACAGTACTAG
GQ131023 63 TATAGGTTAT CATGCGAACA ATTCAACAGA CACTGTAGAC ACAGTACTAG
AB013810 201 Ttgcaccaat taaaat---- ---------- --------AC AAATATCCAG
AB043499 63 TATAGGCTAC CATGCGAACA ACTCAACTGA CACTGTTGAC ACAGTACTTG
AF144305 81 CATTGGTTAC CATGCAAACA ACTCGACAGA gcagGTTGAC ACAATAATGG8888888888 8888888888 8577777777 6666888888 8888888888
FJ981612 113 AAAAGAATGT AACAGTAACA CACTCTGTTA ACCTTCTAGA AGACAAGCAT
GQ117112 113 AAAAGAATGT AACAGTAACA CACTCTGTTA ACCTTCTAGA AGACAAGCAT
GQ131023 113 AAAAGAATGT AACAGTAACA CACTCTGTTA ACCTTCTAGA AGACAAGCAT
AB013810 229 CACTGAACGT GACTATGCCA AACAATGACA A--------- ----------
AB043499 113 AGAAGAACGT GACAGTGACA CACTCTGTCA ACCTACTTGA GGACAGTCAC
AF144305 131 AAAAGAACGT TACTGTTACA CAtgcccaag ACATACTGGA AAAGACACAC8888888677 7777777777 7766666666 6666666666 6666655555
FJ981612 163 AACGGGAAAC TATGCAAACT AAGAGGGGTA GCCCCATTGC ATTTGGGTAA
GQ117112 163 AACGGGAAAC TATGCAAACT AAGAGGGGTA GCCCCATTGC ATTTGGGTAA
GQ131023 163 AACGGGAAAC TATGCAAACT AAGAGGGGTA GCCCCATTGC ATTTGGGTAA
AB013810 260 ---------- ---------- ---------- ---------- ----------
AB043499 163 AACGGAAAAC TATGCCGACT AAAAGGaacA GCCCCACTAC AATTGGGTAA
AF144305 181 AATGGGAAGC TCTGCGATCT AAAtggagtg aagcctctca ttttgagagA5555555555 5555555555 5555554445 5555555555 5555555555
FJ981612 213 ATGTAACATT GCTGGCTGGA TCCTGGGAAA TCCAGAGTGT GAATCACTCT
GQ117112 213 ATGTAACATT GCTGGCTGGA TCCTGGGAAA TCCAGAGTGT GAATCACTCT
GQ131023 213 ATGTAACATT GCTGGCTGGA TCCTGGGAAA TCCAGAGTGT GAATCACTCT
AB013810 260 ---------- ---------- ---------- ---------- ----------
AB043499 213 TTGCAGCATT GCCGGATGGA TCTTAGGAAA TCCAGAATGC GAATCACTgt
AF144305 231 TTGTAGTGTA GCTGGATGGC TCCTCGGAAA CCCTATGTGT Gacgaattca5666666666 6666656666 6666666666 6666666666 6666666644
FJ981612 263 CCACAGCAAG CTCATGGTCC TACATTGTGG AAACATCTAG TTCAGACAAT
GQ117112 263 CCACAGCAAG CTCATGGTCC TACATTGTGG AAACATCTAG TTCAGACAAT
GQ131023 263 CCACAGCAAG CTCATGGTCC TACATTGTGG AAACATCTAG TTCAGACAAT
AB013810 260 ---------- ---------- ---------- ---------- ----------
AB043499 263 tttctaagga aTCATGGTCT TACATTGCAG AAACAccaaa ccctaaaAAT
AF144305 281 tcaatgtgcc ggaATGGTCT TACATAGTGG AGAAGGCCAG TCCAGCCAAT4444444444 4556666666 6666666666 6666655555 5555555777
FJ981612 313 GGAACGTGTT ACCCAGGAGA TTTCATCGAT TATGAGGAGC TAAGAGAGCA
GQ117112 313 GGAACGTGTT ACCCAGGAGA TTTCATCGAT TATGAGGAGC TAAGAGAGCA
GQ131023 313 GGAACGTGTT ACCCAGGAGA TTTCATCGAT TATGAGGAGC TAAGAGAGCA
AB013810 260 ---------- ---------- ---------- ---------- ----------
AB043499 313 GGAACATGTT ACCCAGGGTA TTTCGCCGAC TATGAGGAAC TGAGGGAGCA
AF144305 331 GacctcTGTT ACCCAGGGGA TTTCAACGAC TATGAAGAAC TGAaacacct7666668888 8888888888 8888888888 8888888668 8776666666
FJ981612 363 ATTGAGCTCA GTGTCATCAT TTGAAAGGTT TGAGATATTC CCCAAGACAA
GQ117112 363 ATTGAGCTCA GTGTCATCAT TTGAAAGGTT TGAGATATTC CCCAAGACAA
GQ131023 363 ATTGAGCTCA GTGTCATCAT TTGAAAGGTT TGAGATATTC CCCAAGACAA
AB013810 260 ---------- ---------- -------ATT TGACAAATTg taca------
AB043499 363 ATTGAGCTCA GTATCATCAT TCGAGAGATT TGAAATATTC CCCAAGGATA
AF144305 381 attgagcaga acaaaccatT TTGAGAAAAT TCAGATCATC CCCAA---AA6666666666 6666666666 6666666466 6666666666 6666666666
FJ981612 413 GTTCATGGCC CAATCATGAC TCGAACAAAG GTGTAACGGC AGCATGTCCT
GQ117112 413 GTTCATGGCC CAATCATGAC TCGAACAAAG GTGTAACGGC AGCATGTCCT
GQ131023 413 GTTCATGGCC CAATCATGAC TCGAACAAAG GTGTAACGGC AGCATGTCCT
AB013810 277 ---------- ---------- ---------- ---------- ----------
AB043499 413 GCTCATGGCC CAACCAcact gtaACCAAAG GAGTGACGGC ATCATGCTCC
AF144305 428 GTTCTTGGTC CAATCATGAt gcctcatcAG GGGTGAGCTC AGCATGTCCA6666666666 6666665554 4445555555 5555555555 5555555555
FJ981612 463 CATGCTGGAG CAAAAAGCTT CTACAAAAAT TTAATATGGC TAGTTAAAAA
GQ117112 463 CATGCTGGAG CAAAAAGCTT CTACAAAAAT TTAATATGGC TAGTTAAAAA
GQ131023 463 CATGCTGGAG CAAAAAGCTT CTACAAAAAT TTAATATGGC TAGTTAAAAA
AB013810 277 ---------- ---------- ---------- ---------- ----------
AB043499 463 CATAATGGGA aaagcAGCTT TTACAAAAAT TTGCTATGGC TGACGGAGAA
AF144305 478 TACCATGGGA ggtcctcCTT TTTCAGAAAT GTGGTATGGC TTATCAAAAA5555555544 4444455666 6666666666 6666666666 6555555554
FJ981612 513 AGGAAATTCA TACCCAAAGC TCAGCAAATC CTACATTAAT GATAAAGGGA
GQ117112 513 AGGAAATTCA TACCCAAAGC TCAGCAAATC CTACATTAAT GATAAAGGGA
GQ131023 513 AGGAAATTCA TACCCAAAGC TCAGCAAATC CTACATTAAT GATAAAGGGA
AB013810 277 ---------- ---------- ---------- ---------- ----------
AB043499 513 GAAtggcttg TACCCAAATC TGAGCAAGTC CTATGTAAAC AAAAAGGGaA
AF144305 528 GAAcAGTGCA TACCCAACAA TAAAGAGGAG CTACAATAAT accaaccaag4444444444 5555555555 5555555555 5555555555 5555555546
FJ981612 563 AAGAAGTCCT CGTGCTATGG GGCATTCACC ATCCATCTAC TAGTGCTGAC
GQ117112 563 AAGAAGTCCT CGTGCTATGG GGCATTCACC ATCCATCTAC TAGTGCTGAC
GQ131023 563 AAGAAGTCCT CGTGCTATGG GGCATTCACC ATCCATCTAC TAGTGCTGAC
AB013810 277 ---------- -----TTTGG GGGGTTCACC ACCCGAGTAC GGACAGTGAC
AB043499 563 AAGAAGTCCT TGTGCTATGG GGTGTTCATC ACCCGTCTAa catgggggac
AF144305 578 AAGATCTTTT AGTACTGTGG GGGATTCACC ATCCtaatga tgcggcagag7777777777 7777777777 7777777777 7777766664 4444444444
FJ981612 613 CAA------- ---------- ---------- -----CAAAG TCTCTATCAG
GQ117112 613 CAA------- ---------- ---------- -----CAAAG TCTCTATCAG
GQ131023 613 CAA------- ---------- ---------- -----CAAAG TCTCTATCAG
AB013810 312 CAAaccagcc tatatgctca agcatcaggg agagtCACAG TCTCTACCAA
AB043499 613 caacgggcca ---------- ---------- ---------- --TCTATCAT
AF144305 628 cagacaaag- ---------- ---------- ---------- -CTCTATCAA4440000000 0000000000 0000000000 0000044444 4455555555
FJ981612 631 AATGCAGATG CATATGTTTT TGTGGGGTCA TCAAGATACA GCAAGAAGTT
GQ117112 631 AATGCAGATG CATATGTTTT TGTGGGGTCA TCAAGATACA GCAAGAAGTT
GQ131023 631 AATGCAGATG CATATGTTTT TGTGGGGTCA TCAAGATACA GCAAGAAGTT
AB013810 362 AAgaagccaa caaactg--- ---------- ---------- ---------T
AB043499 631 AAAGAAAATG CTTATGTTTC TGTATTGTCT TCacattata gcagaAGATT
AF144305 646 AACCCAACCA CTTACATTTC CGTTGGAACA Tcaacactga accagAGATT5555555555 5555555555 5555555555 5544444444 4444444466
FJ981612 681 CAAGCCGGAA ATAGCAATAA GACCCAAAGT GAGGGATCAA GAAGGGAGAA
GQ117112 681 CAAGCCGGAA ATAGCAATAA GACCCAAAGT GAGGGATCAA GAAGGGAGAA
GQ131023 681 CAAGCCGGAA ATAGCAATAA GACCCAAAGT GAGGGATCAA GAAGGGAGAA
AB013810 380 AATCCCGAAT ATCGGATCTA GACCCTGGGT AAGGGGTgtc tccaGCAGAA
AB043499 681 CACCCCAGAA ATAGCAAAAA GGCCCAAAGT AAGAGATCAA GAAGGGAGAA
AF144305 696 GGTTCCAGAA ATAGCTACTA GACCCAAAGT AAACGGgCAA AGTGGAAGAA6666777777 7777777777 7777777777 6654666666 6667779999
FJ981612 731 TGAACTATTA CTGGACACTA GTAGAGCCGG GAGACAAAAT AACATTCGAA
GQ117112 731 TGAACTATTA CTGGACACTA GTAGAGCCGG GAGACAAAAT AACATTCGAA
GQ131023 731 TGAACTATTA CTGGACACTA GTAGAGCCGG GAGACAAAAT AACATTCGAA
AB013810 430 TAAGCATCTA TTGGACAATA GTAAAACCGG GAGACAtact tctgattaAC
AB043499 731 TTAACTACTA CTGGACTCTG CTGGAACCCG GGGACACAAT AATATTTGAG
AF144305 746 TGGAGTTCTT CTGGACAATT TTAAAGCCGa atgatgCCAT CAATTTCGAG9999999999 9999998886 6666668877 7777776666 6666666666
FJ981612 781 GCAACTGGAA ATCTAGTGGT ACCGAGATAT GCATTCGCAA TGGAAAGAAA
GQ117112 781 GCAACTGGAA ATCTAGTGGT ACCGAGATAT GCATTCGCAA TGGAAAGAAA
GQ131023 781 GCAACTGGAA ATCTAGTGGT ACCGAGATAT GCATTCGCAA TGGAAAGAAA
AB013810 480 AGCACAGGGA ATCTAATTGC TCCTCGGGGT TACTTCAAAA Tacgaagtgg
AB043499 781 GCAAATGGAA ATCTAATAGC GCCGTGGTAC GCTTTCGCAC TGAGTAGAgg
AF144305 796 AGTAATGGAA ATTTCATTGC TCCAGAATAT GCATACAAAA Ttgtcaagaa6666666666 6666644444 4444444444 4444444444 4444444444
FJ981612 831 TGCTGGATCT GGTATTATCA TTTCAGATAC ACCAGTCCAC GATTGCAATA
GQ117112 831 TGCTGGATCT GGTATTATCA TTTCAGATAC ACCAGTCCAC GATTGCAATA
GQ131023 831 TGCTGGATCT GGTATTATCA TTTCAGATAC ACCAGTCCAC GATTGCAATA
AB013810 530 gaaaagctc- --AATAATGA GGTCAGATGC ACCCATTGGC AAATGCAATT
AB043499 831 cttTGGGTCA GGAATCATCA TCTCAAACGC ATCAATGGGT GAATGTGACG
AF144305 846 aggggactca gcaattatga aaagtgaatt ggaataTGGT AACTGCAACA4444444444 4445555555 5555555555 5555554444 4445555555
FJ981612 881 CAACTTGTCA GACACCCAAG GGTGCTATAA ACACCAGCCT CCCATTTCAG
GQ117112 881 CAACTTGTCA GACACCCAAG GGTGCTATAA ACACCAGCCT CCCATTTCAG
GQ131023 881 CAACTTGTCA GACACCCAAG GGTGCTATAA ACACCAGCCT CCCATTTCAG
AB013810 577 CTGAATGCAT CACTCCAAAT GGaagcattc ccaatgaaaa aCCATTTCAA
AB043499 881 CTAAGTGTCA AACACCCCAA GGAGCTATAA ACAGTAGTCT CCCCTTCCAG
AF144305 896 CCAAGTGTCA AACTCCAATG GGGGCGATAA ACTCTAGTAT GCCATTCCAC5555566666 6666666666 6666666666 6654466666 6777777777
FJ981612 931 AATATACATC CGATCACAAT TGGAAAATGT CCAAAATATG TAAAAAGCAC
GQ117112 931 AATATACATC CGATCACAAT TGGAAAATGT CCAAAATATG TAAAAAGCAC
GQ131023 931 AATATACATC CGATCACAAT TGGAAAATGT CCAAAATATG TAAAAAGCAC
AB013810 627 AATGTAAACA GGATCACATA TGGGGCCTGT CCCAGATATG TTAAGCAAAA
AB043499 931 AATGTACACC CAGTCACAAT AGGAGAGTGT CCAAAGTATG TCAGGAGTAC
AF144305 946 AACATACACC CCCTCACCAT CGGGGAATGC CCCAAATATG TGAAATCAAA7777777777 7777777666 6666555666 6666666666 6666655555
FJ981612 981 AAAATTGAGA CTGGCCACAG GATTGAGGAA TGTCCCGTCT ATTCAATCT-
GQ117112 981 AAAATTGAGA CTGGCCACAG GATTGAGGAA TGTCCCGTCT ATTCAATCT-
GQ131023 981 AAAATTGAGA CTGGCCACAG GATTGAGGAA TGTCCCGTCT ATTCAATCT-
AB013810 677 CACtcTGAAA TTGGCAACAG GGATGCGGAA TGTa------ ----------
AB043499 981 AAAATTAAGG ATGGTTACAG GACTAAGGAA CGTCCCATCC ATTCAATCC-
AF144305 996 CAGATTAGTC CTTGCGACTG GACTCAGAAA TACCCCtcag agagagagaa5555566666 6666556666 6666666666 6655555555 5555555550
FJ981612 1030 ---------- -AGAGGCCTA TTTGGGGCCA TTGCCGGTTT CATTGAAGGG
GQ117112 1030 ---------- -AGAGGCCTA TTTGGGGCCA TTGCCGGTTT CATTGAAGGG
GQ131023 1030 ---------- -AGAGGCCTA TTTGGGGCCA TTGCCGGTTT CATTGAAGGG
AB013810 711 ---------- ---------- ---------- ---------- ----------
AB043499 1030 ---------- -AGA------ ---------- ---------- ----------
AF144305 1046 gaagaaaaaa gAGAGGACTA TTTGGAGCTA TAGCAGGTTT TATAGAGGGa0000000000 0777555555 5555555555 5555555555 5555555554
FJ981612 1069 GGGTGGACAG GGATGGTAGA TGGATGGTAC GGTTATCACC ATCAAAATGA
GQ117112 1069 GGGTGGACAG GGATGGTAGA TGGATGGTAC GGTTATCACC ATCAAAATGA
GQ131023 1069 GGGTGGACAG GGATGGTAGA TGGATGGTAC GGTTATCACC ATCAAAATGA
AB013810 711 ---------- ---------- ---------- ---------- ----------
AB043499 1033 ---------- ---------- ---------- ---------- ----------
AF144305 1096 ggatggcagG GAATGGTAGA TGGTTGGTAT GGGTACCACC ATagcAATGA4444444445 5555555555 5555555555 5555555555 5544455555
FJ981612 1119 GCAGGGGTCA GGATATGCAG CCGACCTGAA GAGCACACAG AATGCCATTG
GQ117112 1119 GCAGGGGTCA GGATATGCAG CCGACCTGAA GAGCACACAG AATGCCATTG
GQ131023 1119 GCAGGGGTCA GGATATGCAG CCGACCTGAA GAGCACACAG AATGCCATTG
AB013810 711 ---------- ---------- ---------- ---------- ----------
AB043499 1033 ---------- ---------- ---------- ---------- ----------
AF144305 1146 GCAGGGGAGT GGATACGCTG CAGACaaaga atcCACTCAA AAGGCAATAG5555555555 5555555555 5555544444 4444444444 4444444444
FJ981612 1169 ACGAAATTAC TAACAAAGTA AATTCTGTTA TTGAAAAGAT GAATACACAG
GQ117112 1169 ACGAGATTAC TAACAAAGTA AATTCTGTTA TTGAAAAGAT GAATACACAG
GQ131023 1169 ACGAGATTAC TAACAAAGTA AATTCTGTTA TTGAAAAGAT GAATACACAG
AB013810 711 ---------- ---------- ---------- ---------- ----------
AB043499 1033 ---------- ---------- ---------- ---------- ----------
AF144305 1196 ATGGAGTCAC CAATAAGGTC AACTCGATCA TTGACAAAAT GAACACTCAG4444444444 4444444444 4444444444 4455555555 5555555555
FJ981612 1219 TTCACAGCAG TAGGTAAAGA GTTCAACCAC CTGGAAAAAA GAATAGAGAA
GQ117112 1219 TTCACAGCAG TAGGTAAAGA GTTCAACCAC CTGGAAAAAA GAATAGAGAA
GQ131023 1219 TTCACAGCAG TAGGTAAAGA GTTCAACCAC CTGGAAAAAA GAATAGAGAA
AB013810 711 ---------- ---------- ---------- ---------- ----------
AB043499 1033 ---------- ---------- ---------- ---------- ----------
AF144305 1246 TTTGAGGCCG TTGGaaggGA ATTTAATAAC TTGGAAAGGA GGATAGAGAA5444444444 4444444455 5555555555 5555555555 5555555555
FJ981612 1269 TTTAAATAAA AAAGTTGATG ATGGTTTCCT GGACATTTGG ACTTACAATG
GQ117112 1269 TTTAAATAAA AAAGTTGATG ATGGTTTCCT GGACATTTGG ACTTACAATG
GQ131023 1269 TTTAAATAAA AAAGTTGATG ATGGTTTCCT GGACATTTGG ACTTACAATG
AB013810 711 ---------- ---------- ---------- ---------- ----------
AB043499 1033 ---------- ---------- ---------- ---------- ----------
AF144305 1296 TTTAAAcaag cagatggaaG ACGGATTCCT AGATGTCTGG ACTTATAATG5555554444 4444444445 5555555555 5555555555 5555555555
FJ981612 1319 CCGAACTGTT GGTTCTATTG GAAAATGAAA GAACTTTGGA CTACCACGAT
GQ117112 1319 CCGAACTGTT GGTTCTATTG GAAAATGAAA GAACTTTGGA CTACCACGAT
GQ131023 1319 CCGAACTGTT GGTTCTATTG GAAAATGAAA GAACTTTGGA CTACCACGAT
AB013810 711 ---------- ---------- ---------- ---------- ----------
AB043499 1033 ---------- ---------- ---------- ---------- ----------
AF144305 1346 CTGAACTtcT GGTTCTCATG GAAAATGAGA GAACTCTAGA CTttCATGAC5555555445 5555555555 5555555555 5555555555 5544555555
FJ981612 1369 TCAAATGTGA AGAACTTATA TGAAAAGGTA AGAAGCCAGC TAAAAAACAA
GQ117112 1369 TCAAATGTGA AGAACTTATA TGAAAAGGTA AGAAGCCAGT TAAAAAACAA
GQ131023 1369 TCAAATGTGA AGAACTTATA TGAAAAGGTA AGAAGCCAGC TAAAAAACAA
AB013810 711 ---------- ---------- ---------- ---------- ----------
AB043499 1033 ---------- ---------- ---------- ---------- ----------
AF144305 1396 TCAAATGTCA AGAACCTTTA TGACAAGGTc cgactaCAGC TTAGGGATAA5555555555 5555555555 5555555554 4444444444 4444444444
FJ981612 1419 TGCCAAGGAA ATTGGAAACG GCTGCTTTGA ATTTTACCAC AAATGCGATA
GQ117112 1419 TGCCAAGGAA ATTGGAAACG GCTGCTTTGA ATTTTACCAC AAATGCGATA
GQ131023 1419 TGCCAAGGAA ATTGGAAACG GCTGCTTTGA ATTTTACCAC AAATGCGATA
AB013810 711 ---------- ---------- ---------- ---------- ----------
AB043499 1033 ---------- ---------- ---------- ---------- ----------
AF144305 1446 TGCAAAGGAg cTGGGTAATG GTTGTTTCGA GTTCTATCAC AAATGTGATA4444444444 4555555555 5555555555 5555555555 5555555555
FJ981612 1469 ACACGTGCAT GGAAAGTGTC AAAAATGGGA CTTATGACTA CCCAAAATAC
GQ117112 1469 ACACGTGCAT GGAAAGTGTC AAAAATGGGA CTTATGACTA CCCAAAATAC
GQ131023 1469 ACACGTGCAT GGAAAGTGTC AAAAATGGGA CTTATGACTA CCCAAAATAC
AB013810 711 ---------- ---------- ---------- ---------- ----------
AB043499 1033 ---------- ---------- ---------- ---------- ----------
AF144305 1496 AtgaaTGTAT GGAAAGTGTA AAAAACGGAA CGTATGACTA CCCgcagTAT5444466666 6666666666 6666666666 6666666666 6664444666
FJ981612 1519 TCAGAGGAAG CAAAATTAAA CAGAGAAGAA ATAGATGGGG TAAAACTGGA
GQ117112 1519 TCAGAGGAAG CAAAATTAAA CAGAGAAGAA ATAGATGGGG TAAAGCTGGA
GQ131023 1519 TCAGAGGAAG CAAAATTAAA CAGAGAAGAA ATAGATGGGG TAAAGCTGGA
AB013810 711 ---------- ---------- ---------- ---------- ----------
AB043499 1033 ---------- ---------- ---------- ---------- ----------
AF144305 1546 TCAGAAGAAG CAAGACTAAA CAGAGAGGAA ATAagTGGAG TAAAATTGGA6666666666 6666666666 6666666666 6664455555 5522244444
FJ981612 1569 ATCAACAAGG ATTTACCAGA TTTTGGCGAT CTATTCAACT GTCGCCAGTT
GQ117112 1569 ATCAACAAGG ATTTACCAGA TTTTGGCGAT CTATTCAACT GTCGCCAGTT
GQ131023 1569 ATCAACAAGG ATTTACCAGA TTTTGGCGAT CTATTCAACT GTCGCCAGTT
AB013810 711 ---------- ---------- ---------- ---------- ----------
AB043499 1033 ---------- ---------- ---------- ---------- ----------
AF144305 1596 ATCAATGGGA ACTTACCAAA TACTGtcaAT TTATTCAACA GTGGCGAGTT4444444444 4445555555 5555544455 5555555555 5555555555
FJ981612 1619 CATTGGTACT GGTAGTCTCC CTGGGGGCAA TCAGTTTCTG GATGTGCTCT
GQ117112 1619 CATTGGTACT GGTAGTCTCC CTGGGGGCAA TCAGTTTCTG GATGTGCTCT
GQ131023 1619 CATTGGTACT GGTAGTCTCC CTGGGGGCAA TCAGTTTCTG GATGTGCTCT
AB013810 711 ---------- ---------- ---------- ---------- ----------
AB043499 1033 ---------- ---------- ---------- ---------- ----------
AF144305 1646 CCCTAGCACT GGCAATCatg gtagctggtc tatcTTTATG GATGTGCTCC5555555555 5555555444 4444444444 4444555555 5555555555
FJ981612 1669 AATGGGTCTC TACAGTGTAG AATATGTATT TAA------- ----------
GQ117112 1669 AATGGGTCTC TACAGTGTAG AATATGTATT TAA------- ----------
GQ131023 1669 AATGGGTCTC TACAGTGTAG GATATGTATT TAA------- ----------
AB013810 711 ---------- ---------- ---------- ---------- ----------
AB043499 1033 ---------- ---------- ---------- ---------- ----------
AF144305 1696 AATGGATCGT TACAATGCAG AATttgcatt taaatttgtg agttcagatt5555555555 5544433333 3332222222 2220000000 0000000000
FJ981612 1702 ---------- -----
GQ117112 1702 ---------- -----
GQ131023 1702 ---------- -----
AB013810 711 ---------- -----
AB043499 1033 ---------- -----
AF144305 1746 gtagttaaaa acacc0000000000 00000
一緒に出力されるFASTA形式のファイルとともに、系統樹作成のためのデータも出力されます。
Sequence tree:
==============Tree constructed using UPGMA based on DIALIGN fragment weight scores
((((FJ981612 :0.000585(GQ117112 :0.000583GQ131023 :0.000583):0.000002)
:0.002449AB043499 :0.003033):0.001340AF144305 :0.004373)
:0.032379AB013810 :0.036752);
これを見ると、GQ117112(豚)-GQ131023(豚)-FJ981612(豚)が1つの塊で、AB043499(ソ連), AF144305(鳥), AB013810(香港)の順で違いが大きくなっている、という計算結果が出ています。鳥インフルの遺伝子配列の方が近いとはちょっと意外ですが。それはソ連型と香港型の遺伝子配列が完全長ではないためかもしれません。
投稿者 ただ : 23:53 | トラックバック (0) カテゴリー ; 生命科学
2009年5月25日
 EMBOSSを試す その6
EMBOSSを試す その6
前回edialignコマンドで出力したマルチプルアライメントのデータのうち、FASTA形式のファイルはshowalignコマンドで別の形式で表示することができます。edialignで出力したFASTA形式のファイルをali.fastaとします。
$ showalign ali.fasta
Display a multiple sequence alignment in pretty format
Output file [ali.showalign]: stdout
10 20 30 40 50 60
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus ---nnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnn
FJ981612 ------------------------------------------------------------
GQ117112 ------------------------------------------------------------
GQ131023 ------------------------------------------------------------
AB013810 TTGTTGAACGCAGCAAAGCTTACAGCAACTGTTACCCTTATGATGTGCCGGATTATGCCT
AB043499 ------------------------------------------------------------
AF144305 GCA---------------------------------------------------------
Consensus ---nnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnn70 80 90 100 110 120
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus nnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnATGAAgGCAAtACTA
FJ981612 ---------------------------------------------...............
GQ117112 ---------------------------------------------...............
GQ131023 ---------------------------------------------...............
AB013810 CCCTTAGGTCACTAGTTGCCTCATCCGGCACCCTGGAGTTTAACA.....A..TTC.A.T
AB043499 ---------------------------------------------.....A....A....
AF144305 ------------------------------------------------------------
Consensus nnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnATGAAgGCAAtACTA130 140 150 160 170 180
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus GTAGTTCTGcTaTaTACnnnnnn--nn----nnnnn------n-n----nATTTgCAaCC
FJ981612 .................---------------------------------..........
GQ117112 .................---------------------------------..........
GQ131023 .................---------------------------------..........
AB013810 .G.C.GGA.TCGCTC.GAATGGAACAAGCTTTGCTTGCAAAAGGAGATCT...AAA.GTT
AB043499 C........T.G.G.G.---------------------------------....A..G.T
AF144305 .GG..ATAATCTGTC.AAATGGAGAAAATAGTGCTTCTTCTTGCAATAGTCAG.CTTGT.
Consensus GTAGTTCTGcTaTaTACnnnnnn--nn----nnnnn------n-n----nATTTgCAaCC190 200 210 220 230 240
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus gCAAATGCAGACACAtTATGTATAGGTTATCATGCGAACAAtTCAACAGACACTGTaGAC
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 T.TTTA.T...TTG.A.TG..TGCACCA..T.AAAT----------------------..
AB043499 A..T...........A..........C..C...........C.....T........T...
AF144305 AA..G..ATC.G---A.T..C..T.....C.....A.....C..G.....GCAG..T...
Consensus gCAAATGCAGACACAtTATGTATAGGTTATCATGCGAACAAtTCAACAGACACTGTaGAC250 260 270 280 290 300
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus ACAGTACTAGAAAAGAAtGTaACAGTaACACACTCTGTtAACCTtCTaGAAGACAagCAt
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 .A.TAT.C..C.CT...C..G..TA.GC..A..AA..AC..-------------------
AB043499 ........T..G.....C..G.....G...........C.....A..T..G....GT..C
AF144305 ...A..A.G........C..T..T..T.....TG.CCAAG..A.A..G...A.G.CA..C
Consensus ACAGTACTAGAAAAGAAtGTaACAGTaACACACTCTGTtAACCTtCTaGAAGACAagCAt310 320 330 340 350 360
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus AACGGGAAACTATGCaAACTAAgAGGgGTAGCCCCAtTgCATTTGGGTAAaTGTAaCATT
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 ------------------------------------------------------------
AB043499 .....A.........CG.....A...AAC.......C.A..A........T..C.G....
AF144305 ..T.....G..C...G.T....AT..A..GAAG..TC.CAT....A.AG.T....GTG.A
Consensus AACGGGAAACTATGCaAACTAAgAGGgGTAGCCCCAtTgCATTTGGGTAAaTGTAaCATT370 380 390 400 410 420
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus GCTGGcTGGATCCTgGGAAATCCAGAGTGTGAATCACTCTcCACaGcaagcTCATGGTCc
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 ------------------------------------------------------------
AB043499 ..C..A......T.A...........A..C........G.TTT.TAAGGAA........T
AF144305 .....A...C....C.....C..TAT......CGA.T..AT..AT.TGCCGGA......T
Consensus GCTGGcTGGATCCTgGGAAATCCAGAGTGTGAATCACTCTcCACaGcaagcTCATGGTCc430 440 450 460 470 480
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus TACATTGTGGAAACAtCtAGTtCAGACAATGGAACgTGTTACCCAGGaGATTTCAtCGAt
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 ------------------------------------------------------------
AB043499 .......CA......C.A.ACC.TA.A........A...........GT.....GC...C
AF144305 .....A.....G.AGG.C...C...C.....ACCTC...........G.......A...C
Consensus TACATTGTGGAAACAtCtAGTtCAGACAATGGAACgTGTTACCCAGGaGATTTCAtCGAt490 500 510 520 530 540
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus TATGAGGAgCTaAGAGAGCAATTGAGCTCAGTgTCATCATTTGAaAGgTTTGAGATATTC
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 -----------------------------------------------A.....C.A...G
AB043499 ........A..G..G.................A........C..G..A.....A......
AF144305 .....A..A..G.A.C.C.T.......AG.ACAAACCAT.....G.AAA..C....CA..
Consensus TATGAGGAgCTaAGAGAGCAATTGAGCTCAGTgTCATCATTTGAaAGgTTTGAGATATTC550 560 570 580 590 600
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus CCCAAGacAAGTTCATGGCCCAATCATGActCgAaCAAAGGtGTaACGGCAGCATGTCCt
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 TA..--------------------------------------------------------
AB043499 ......GAT..C...........C..CACTGTA.C......A..G......T....CT.C
AF144305 .....---......T...T..........TG.CTCATC...G..G.GCT..........A
Consensus CCCAAGacAAGTTCATGGCCCAATCATGActCgAaCAAAGGtGTaACGGCAGCATGTCCt610 620 630 640 650 660
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus CATgcTGGagcAAaaAGCTTcTACAAAAATTTaaTATGGCTagTtAAAAAaggaAaTTCA
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 ------------------------------------------------------------
AB043499 ...AA...GAA..GC.....T...........GC.......GACGG.G..GAATGGC.TG
AF144305 T.CCA...GAGGTCCTC...T.T..G....G.GG.......TA.C.....GAAC.G.G..
Consensus CATgcTGGagcAAaaAGCTTcTACAAAAATTTaaTATGGCTagTtAAAAAaggaAaTTCA670 680 690 700 710 720
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus TACCCAAAgCTcAGCAAaTCCTACATTAATgAtAAaGGgAAAGAAGTCCTcGTGCTATGG
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 -------------------------------------------------------.T...
AB043499 ........T..G.....G.....TG.A..CA.A..G..A...........T.........
AF144305 .......CAA.A.AG.GGAG.....A....ACC..CCAAG....TC.TT.A..A..G...
Consensus TACCCAAAgCTcAGCAAaTCCTACATTAATgAtAAaGGgAAAGAAGTCCTcGTGCTATGG730 740 750 760 770 780
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus GGcATTCACCATCCaTCTACTAgtGCTGACCAAnn-n-n-nnnnnnnnnnnnnnnnnnnn
FJ981612 .................................---------------------------
GQ117112 .................................---------------------------
GQ131023 .................................---------------------------
AB013810 ..GG.......C..GAG...GGACAG.......ACCAGCCTATATGCTCAAGCATCAGGG
AB043499 ..TG....T..C..G....AC.TG.GG......CGGGCCA--------------------
AF144305 ..G...........TAA.GA.GCG..A..G..GACAAAG---------------------
Consensus GGcATTCACCATCCaTCTACTAgtGCTGACCAAnn-n-n-nnnnnnnnnnnnnnnnnnnn790 800 810 820 830 840
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus nnnnnCAaAGTCTCTATCAgAAtGCAgATGCATATGTTTtTGTgGGGTCATCAAgATAcA
FJ981612 -----.......................................................
GQ117112 -----.......................................................
GQ131023 -----.......................................................
AB013810 AGAGT..C........C..A..GAAGCCAA..A.CTG-----------------------
AB043499 ------------.......T..A.A.A....T.......C...ATT...T...CAT..T.
AF144305 -----------........A..CC..ACCA.T..CA...CC..T..AA......C.CTG.
Consensus nnnnnCAaAGTCTCTATCAgAAtGCAgATGCATATGTTTtTGTgGGGTCATCAAgATAcA850 860 870 880 890 900
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus GCAAGAagTTCAagCCGGAAATAGCAAtAAGACCCAAAGTgAGGGATCAAGAAGGGAGAA
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 ---------.A.TC...A.T..C.G.TCT......TGG..A....G.GTCTCCA.C....
AB043499 ...GA.GA....CC..A..........A...G........A..A................
AF144305 A.C...GA..GGTT..A........T.CT...........A.AC.GG...AGT..A....
Consensus GCAAGAagTTCAagCCGGAAATAGCAAtAAGACCCAAAGTgAGGGATCAAGAAGGGAGAA910 920 930 940 950 960
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus TGAACTAtTACTGGACACTAGTAGAGCCGGGAGACAaAATAAcATTCGAaGCAACTGGAA
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 .A.G.ATC..T......A.....A.A..........T.C.TCTGA.TA.CAGC..A..G.
AB043499 .T.....C........T..GC.G..A..C..G....C.....T...T..G....A.....
AF144305 ..G.G.TC.T.......A.TT..A.....AAT..TGCC..C.AT.....GAGT.A.....
Consensus TGAACTAtTACTGGACACTAGTAGAGCCGGGAGACAaAATAAcATTCGAaGCAACTGGAA970 980 990 1000 1010 1020
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus ATCTAgTgGtaCCGaGATATGCATTCGCAATGGaAAGAAAtGcTGGaTCtGGtATTATCA
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 .....A.T.CT..TC.GGG.TAC...AA...ACG...TGGGAAAA.C..---A..A..G.
AB043499 .....A.A.CG...T.G..C..T......C..AGT...GGCTT...G..A..A..C....
AF144305 ..T.CA.T.CT..AGA........A.AA...T.TC.AG..A.GG.AC..A.CA.....G.
Consensus ATCTAgTgGtaCCGaGATATGCATTCGCAATGGaAAGAAAtGcTGGaTCtGGtATTATCA1030 1040 1050 1060 1070 1080
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus TtTCAGATaCACCAgTccaCGAtTGCAATACaActTGTCAgACACCCAAGGGtGCTATAA
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 GG......G....CA.TGG.A.A......T.TGAA..CATC..T..A..T..AAGC..TC
AB043499 .C...A.CG..T..A.GGGT..A..TG.CG.T.AG.....A......C.A..A.......
AF144305 AAAGT..ATTGGA.TATGGTA.C.....C..C.AG.....A..T..A.T...G..G....
Consensus TtTCAGATaCACCAgTccaCGAtTGCAATACaActTGTCAgACACCCAAGGGtGCTATAA1090 1100 1110 1120 1130 1140
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus ACACcAGcCTCCCATTTCAGAATATACAtCCGATCACAATTGGAaAATGTCCAAAATATG
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 C..ATGAAAAA........A...G..A.CAG.......TA...GGCC.....C.G.....
AB043499 ...GT..T.....C..C......G....C..AG.......A...G.G........G....
AF144305 ..T.T..TA.G.....C..C..C.....C..CC....C..C..GG....C..C.......
Consensus ACACcAGcCTCCCATTTCAGAATATACAtCCGATCACAATTGGAaAATGTCCAAAATATG1150 1160 1170 1180 1190 1200
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus TaAAAAGcACAAAATTGAGACTGGCcACAGGAtTGAGGAATGTCCCgTCtATTCAATCtn
FJ981612 ...........................................................-
GQ117112 ...........................................................-
GQ131023 ...........................................................-
AB013810 .T..GCAA.AC.CTC...A.T....A.....GA..C.......A----------------
AB043499 .C.GG..T........A..GA...TT......C.A.....C.....A..C........C-
AF144305 .G...TCA.AC.G...AGTC..T..G..T...C.C..A...AC...TCAG.GAG.GAGAA
Consensus TaAAAAGcACAAAATTGAGACTGGCcACAGGAtTGAGGAATGTCCCgTCtATTCAATCtn1210 1220 1230 1240 1250 1260
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus nnnnnnnnnnnAGAGGcCTATTTGGgGCcATtGCcGGTTTcATtGAaGGgGGgTGGacaG
FJ981612 -----------.................................................
GQ117112 -----------.................................................
GQ131023 -----------.................................................
AB013810 ------------------------------------------------------------
AB043499 -----------...----------------------------------------------
AF144305 GAAGAAAAAAG.....A........A..T..A..A.....T..A..G..A..A...CAG.
Consensus nnnnnnnnnnnAGAGGcCTATTTGGgGCcATtGCcGGTTTcATtGAaGGgGGgTGGacaG1270 1280 1290 1300 1310 1320
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus GgATGGTAGATGGaTGGTAcGGtTAtCACCATcaaAATGAGCAGGGGtcaGGATAtGCaG
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 ------------------------------------------------------------
AB043499 ------------------------------------------------------------
AF144305 .A...........T.....T..G..C......AGC............AGT.....C..T.
Consensus GgATGGTAGATGGaTGGTAcGGtTAtCACCATcaaAATGAGCAGGGGtcaGGATAtGCaG1330 1340 1350 1360 1370 1380
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus CcGACctgaAgagCACaCAgAAtGCcATtGAcGanaTtACtAAcAAaGTaAAtTCtgTtA
FJ981612 ..................................A.........................
GQ117112 ..................................G.........................
GQ131023 ..................................G.........................
AB013810 ------------------------------------------------------------
AB043499 ------------------------------------------------------------
AF144305 .A...AAAG.ATC...T..A..G..A..A..T.GAG.C..C..T..G..C..C..GA.C.
Consensus CcGACctgaAgagCACaCAgAAtGCcATtGAcGanaTtACtAAcAAaGTaAAtTCtgTtA1390 1400 1410 1420 1430 1440
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus TTGAaAAgATGAAtACaCAGTTcacaGCaGTaGGtAaaGAgTTcAAccACcTGGAAAaaA
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 ------------------------------------------------------------
AB043499 ------------------------------------------------------------
AF144305 ....C..A.....C..T.....TGAG..C..T..A.GG..A..T..TA..T......GG.
Consensus TTGAaAAgATGAAtACaCAGTTcacaGCaGTaGGtAaaGAgTTcAAccACcTGGAAAaaA1450 1460 1470 1480 1490 1500
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus GaATAGAGAATTTAAAtAAaaAagTtGAtGAtGGtTTCCTgGAcaTtTGGACTTAcAATG
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 ------------------------------------------------------------
AB043499 ------------------------------------------------------------
AF144305 .G..............C..GC.GA.G..A..C..A.....A..TG.C........T....
Consensus GaATAGAGAATTTAAAtAAaaAagTtGAtGAtGGtTTCCTgGAcaTtTGGACTTAcAATG1510 1520 1530 1540 1550 1560
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus CcGAACTgtTGGTTCTatTGGAAAATGAaAGAACTtTgGACTacCAcGAtTCAAATGTgA
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 ------------------------------------------------------------
AB043499 ------------------------------------------------------------
AF144305 .T.....TC.......CA..........G......C.A....TT..T..C........C.
Consensus CcGAACTgtTGGTTCTatTGGAAAATGAaAGAACTtTgGACTacCAcGAtTCAAATGTgA1570 1580 1590 1600 1610 1620
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus AGAACtTaTATGAaAAGGTaaGAagcCAGcTaAaaaAcAATGCcAAGGAaaTtGGaAAcG
FJ981612 ............................................................
GQ117112 .............................T..............................
GQ131023 ............................................................
AB013810 ------------------------------------------------------------
AB043499 ------------------------------------------------------------
AF144305 .....C.T.....C.....CC..CTA.....T.GGG.T.....A.....GC.G..T..T.
Consensus AGAACtTaTATGAaAAGGTaaGAagcCAGcTaAaaaAcAATGCcAAGGAaaTtGGaAAcG1630 1640 1650 1660 1670 1680
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus GcTGcTTtGAaTTtTAcCACAAATGcGATAAcacgTGcATGGAAAGTGTcAAAAAtGGgA
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 ------------------------------------------------------------
AB043499 ------------------------------------------------------------
AF144305 .T..T..C..G..C..T........T.....TGAA..T...........A.....C..A.
Consensus GcTGcTTtGAaTTtTAcCACAAATGcGATAAcacgTGcATGGAAAGTGTcAAAAAtGGgA1690 1700 1710 1720 1730 1740
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus CtTATGACTACCCaaAaTAcTCAGAgGAAGCAAaAtTAAACAGAGAaGAAATAgaTGGgG
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 ------------------------------------------------------------
AB043499 ------------------------------------------------------------
AF144305 .G...........GC.G..T.....A.......G.C..........G......AG...A.
Consensus CtTATGACTACCCaaAaTAcTCAGAgGAAGCAAaAtTAAACAGAGAaGAAATAgaTGGgG1750 1760 1770 1780 1790 1800
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus TAAAncTGGAATCAAcaaGgAtTTACCAgATttTGgCgATcTATTCAACtGTcGCcAGTT
FJ981612 ....A.......................................................
GQ117112 ....G.......................................................
GQ131023 ....G.......................................................
AB013810 ------------------------------------------------------------
AB043499 ------------------------------------------------------------
AF144305 ....AT.........TGG.A.C......A..AC..T.A..T........A..G..G....
Consensus TAAAncTGGAATCAAcaaGgAtTTACCAgATttTGgCgATcTATTCAACtGTcGCcAGTT1810 1820 1830 1840 1850 1860
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus CatTgGtACTGGtAgTCtcccTgGggGcaaTcagTTTcTGGATGTGCTCtAATGGgTCtc
FJ981612 ............................................................
GQ117112 ............................................................
GQ131023 ............................................................
AB013810 ------------------------------------------------------------
AB043499 ------------------------------------------------------------
AF144305 .CC.A.C.....C.A..ATGG.A.CT.GTC.ATC...A...........C.....A..GT
Consensus CatTgGtACTGGtAgTCtcccTgGggGcaaTcagTTTcTGGATGTGCTCtAATGGgTCtc1870 1880 1890 1900 1910 1920
----:----|----:----|----:----|----:----|----:----|----:----|
Consensus TACAgTGtAGaATaTGtATTTAAnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnn
FJ981612 .......................--------------------------------
GQ117112 .......................--------------------------------
GQ131023 ..........G............--------------------------------
AB013810 -------------------------------------------------------
AB043499 -------------------------------------------------------
AF144305 ....A..C.....T..C......ATTTGTGAGTTCAGATTGTAGTTAAAAACACC
Consensus TACAgTGtAGaATaTGtATTTAAnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnnn
投稿者 ただ : 00:49 | トラックバック (0) カテゴリー ; 生命科学
 もうそろそろ梅雨のようで
もうそろそろ梅雨のようで
マスクって、いつまでつけてればいいんですかね。「もうええよ」って誰かが言うわけではなかろうに。終息宣言はもうそろそろ出てもええのかも知れんけども…ワクチンができたわけではないからねぇ。メキシコでも、海沿いの暑い地域ではあんまり流行ってなかったらしいから、こっちでも季節性と同様に梅雨入りと共に小康状態になることだってあるわけで、そうなったとしても、やっぱりマスクをつけてる人は多いままなんだろうかと。というかいつまで心配するんだと。
昨日、I市立病院に行ったんですが、マスク絶対着用なんですねー。まぁ出入り口での消毒(ヒビテン消毒やった)はまぁアリやとしても…。無条件にマスク着用ってなんか「お前らきたねぇから」と言わんばかりの対応よね。強毒性を基準にした対応やからしゃあないんかも知れんけど。季節性インフルなんて全然無防備やん、医者も看護師も。なんか全然違って物騒やなぁ。
投稿者 ただ : 23:04 | トラックバック (0) カテゴリー ; mein Erbe
2009年5月26日
 EMBOSSを試す その7
EMBOSSを試す その7 
塩基配列を扱うときに、制限酵素(切断)地図を出せると組み換えをしたり変異を見つけたり(RFLP)するのに便利です。それがremapコマンドです。ただ…CUIでは環状で出せないのが無念ですね。
$ remap genbank:GQ149630
Display restriction enzyme binding sites in a nucleotide sequence
Comma separated enzyme list [all]:
Minimum recognition site length [4]:
Output file [gq149630.remap]: stdout
GQ149630
Influenza A virus (A/Mexico/4603/2009(H1N1)) segment 4 hemagglutinin
(HA) gene, complete cds.EMBOSS An error in remap.c at line 238:
Cannot locate enzyme file. Run REBASEEXTRACT
早速やってみると、エラーが出ました。最初に酵素のデータを入力しておかなければならないようです。ここにもあるように、http://rebase.neb.com/rebase/rebase.htmlから"proto.数値"と"withrefm.数値"をダウンロードします。FTPを使ってもいけます。
$ wget ftp://ftp.neb.com/pub/rebase/proto.905
$ wget ftp://ftp.neb.com/pub/rebase/withrefm.905
$ rebaseextract
Process the REBASE database for use by restriction enzyme applications
REBASE database withrefm file: withrefm.905
REBASE database proto file: proto.905
$ remap
Display restriction enzyme binding sites in a nucleotide sequence
Input nucleotide sequence(s): genbank:GQ169382
Comma separated enzyme list [all]:
Minimum recognition site length [4]:
Output file [gq169382.remap]: stdout
GQ169382
Influenza A virus (A/Thailand/104/2009(H1N1)) segment 4 hemagglutinin
(HA) gene, complete cds.SpeI
|MaeI FaiI
Hin4II || TspDTI | FaiI
\ \\ \ \ \
gcaaaagcaggggaaaacaaaagcacaaaatgaaggcaatactagtagttctgctatata
10 20 30 40 50 60
----:----|----:----|----:----|----:----|----:----|----:----|
cgttttcgtccccttttgttttcgtgttttacttccgttatgatcatcaagacgatatat
/ /// / /
Hin4II ||SpeI | FaiI
|MaeI FaiI
TspDTIA K A G E N K S T K * R Q Y * * F C Y I
Q K Q G K T K A Q N E G N T S S S A I Y
K S R G K Q K H K M K A I L V V L L Y T
----:----|----:----|----:----|----:----|----:----|----:----|
A F A P S F L L V F H L C Y * Y N Q * I
X L L L P F C F C L I F A I S T T R S Y
C F C P F V F A C F S P L V L L E A I YSetI
| FatI
| |CviAII
| ||FaiI
| ||| NlaIII
CviRI FaiI | ||| | TspEI
| AciI CviRI | FaiI | ||| | | AgsI
\ \ \ \ \ \ \\\ \ \ \
catttgcaaccgcaaatgcagacacattatgtataggttatcatgcgaacaattcaacag
70 80 90 100 110 120
----:----|----:----|----:----|----:----|----:----|----:----|
gtaaacgttggcgtttacgtctgtgtaatacatatccaatagtacgcttgttaagttgtc
/ / / / // / /// / //
CviRI AciI CviRI | |SetI | ||FatI | |AlwNI
| FaiI | |CviAII | TscAI
FaiI | FaiI TspEI
NlaIII AgsIH L Q P Q M Q T H Y V * V I M R T I Q Q
I C N R K C R H I M Y R L S C E Q F N R
F A T A N A D T L C I G Y H A N N S T D
----:----|----:----|----:----|----:----|----:----|----:----|
C K C G C I C V C * T Y T I M R V I * C
V N A V A F A S V N H I P * * A F L E V
M Q L R L H L C M I Y L N D H S C N L LMseI
|HpaI
AlwNI TatI |MjaIV
|SfeI Tsp4CI |HindII
||Tsp4CI |Csp6I || SetI
||| AccI ||RsaI MaeIII || |XbaI
||| |MjaIV ||ScaI | MaeIII || ||MaeI
||| |TscAI ||| MaeI | Tsp4CI || ||Hpy178III
\\\ \\ \\\ \ \ \ \\ \\\
acactgtagacacagtactagaaaagaatgtaacagtaacacactctgttaaccttctag
130 140 150 160 170 180
----:----|----:----|----:----|----:----|----:----|----:----|
tgtgacatctgtgtcatgatcttttcttacattgtcattgtgtgagacaattggaagatc
/ // / /// / / / // //
| || | ||| MaeI | MaeIII |MseI |XbaI
| || | ||TatI MaeIII |SetI Hpy178III
| || | |Csp6I Tsp4CI HindII MaeI
| || | ScaI MjaIV
| || | RsaI HpaI
| || Tsp4CI
| |AccI
| MjaIV
| SfeI
Tsp4CIT L * T Q Y * K R M * Q * H T L L T F *
H C R H S T R K E C N S N T L C * P S R
T V D T V L E K N V T V T H S V N L L E
----:----|----:----|----:----|----:----|----:----|----:----|
V S Y V C Y * F L I Y C Y C V R N V K *
S V T S V T S S F F T V T V C E T L R R
C Q L C L V L F S H L L L V S Q * G E LHin4II FaiI
| BbvII | CviRI CviJI
| | FaiI | | MnlI | BsrDI
| | | MboII | | | DdeI | | CviRI
\ \ \ \ \ \ \ \ \ \ \
aagacaagcataacgggaaactatgcaaactaagaggggtagccccattgcatttgggta
190 200 210 220 230 240
----:----|----:----|----:----|----:----|----:----|----:----|
ttctgttcgtattgccctttgatacgtttgattctccccatcggggtaacgtaaacccat
/ // / // / // /
Hin4II |MboII | |MnlI DdeI |BsrDI CviRI
|BbvII | CviRI CviJI
FaiI FaiIK T S I T G N Y A N * E G * P H C I W V
R Q A * R E T M Q T K R G S P I A F G *
D K H N G K L C K L R G V A P L H L G K
----:----|----:----|----:----|----:----|----:----|----:----|
F V L M V P F * A F * S P Y G W Q M Q T
S S L C L P F S H L S L P T A G N C K P
L C A Y R S V I C V L L P L G M A N P Y...
AlwNI SfeI
|Hpy178III |BsmAI
|| BseGI |Eco31I
|| | SduI || Tsp4CI FaiI
|| | HgiAI || | TscAI | TfiI
|| | | FokI || | | FaiI MseI | HinfI
\\ \ \ \ \\ \ \ \ \ \ \
gtttctggatgtgctctaatgggtctctacagtgtagaatatgtatttaaccataggatt
1690 1700 1710 1720 1730 1740
----:----|----:----|----:----|----:----|----:----|----:----|
caaagacctacacgagattacccagagatgtcacatcttatacataaattggtatcctaa
/ / / / / /// / / / /
AlwNI | BseGI | | ||Eco31I FaiI | FaiI HinfI
| HgiAI | | ||BsmAI MseI TfiI
| SduI | | |SfeI
Hpy178III | | Tsp4CI
| TscAI
FokIV S G C A L M G L Y S V E Y V F N H R I
F L D V L * W V S T V * N M Y L T I G F
F W M C S N G S L Q C R I C I * P * D S
----:----|----:----|----:----|----:----|----:----|----:----|
T E P H A R I P R * L T S Y T N L W L I
L K Q I H E L P D R C H L I H I * G Y S
N R S T S * H T E V T Y F I Y K V M P N
c-
g
X
-E
# Enzymes that cut Frequency Isoschizomers
Acc65I 1 Asp718I
AccI 1 FblI,XmiI
AciI 1 BspACI,SsiI
AflIII 2
AgsI 10
AhaIII 1 DraI
AluBI 6 AluI
AlwNI 2 CaiI
ApoI 5 AcsI,XapI
AsuI 3 Cfr13I,PspPI,Sau96I,AspS9I
AsuII 1 Bpu14I,Bsp119I,BspT104I,BstBI,Csp45I,NspV,SfuI
AvaII 1 Bme18I,Eco47I,SinI,VpaK11BI
BaeI 1
BalI 1 MlsI,MluNI,MscI,Msp20I
BamHI 1
BbvI 3 BseXI,BstV1I,Lsp1109I
BbvII 1 BpiI,BpuAI,BstV2I,BbsI
BccI 8
BceAI 2
BinI 4 AlwI,BspPI,AclWI
BlsI 3
BmgT120I 3
BmsI 1 LweI,SfaNI
BsaAI 1 BstBAI,Ppu21I
BseBI 5 Bst2UI,BstNI,BstOI,MvaI
BseGI 5 BstF5I,BtsCI
BseMII 3
BseRI 1
BsiI 1 BssSI,Bst2BI,BauI
BsiYI 4 Bsc4I,BseLI,BslI,AfiI
BslFI 2 BsmFI,FaqI
BsmAI 5 Alw26I,BstMAI
BsmI 4 BsaMI,Mva1269I,PctI
BspCNI 3
BspHI 1 CciI,PagI,RcaI
BspQI 1 LguI,PciSI,SapI
BsrDI 4 BseMI,Bse3DI
BsrI 7 BseNI,Bse1I,BsrSI
BssKI 6 BstSCI,StyD4I
BstKTI 5
BstXI 1
Cac8I 2 BstC8I
CauII 1 BcnI,BpuMI,NciI,AsuC2I
Cfr10I 1 BsrFI,BssAI,Bse118I
CfrI 1 AcoI,EaeI
ClaI 1 Bsa29I,BseCI,BshVI,BspDI,BspXI,Bsu15I,BsuTUI,BanIII
Csp6I 4 CviQI,RsaNI
CviAII 7
CviJI 19 CviKI-1
CviRI 10 HpyCH4V
DdeI 5 BstDEI,HpyF3I
DpnI 5 MalI
Ecl136II 1 EcoICRI
Eco31I 1 Bso31I,BspTNI,BsaI
Eco57I 1 AcuI
Eco57MI 2
EcoP15I 1
EcoRII 5 AjnI,Psp6I,PspGI
EcoT22I 2 Mph1103I,NsiI,Zsp2I
EspI 1 Bpu1102I,Bsp1720I,CelII,BlpI
FaiI 29
FatI 7
Fnu4HI 3 BisI,Fsp4HI,GluI,ItaI,SatI
FokI 5
GsuI 1 BpmI
HaeIII 4 BsnI,BsuRI,BshFI,PhoI
HgiAI 3 Bbv12I,BsiHKAI,Alw21I
HgiCI 1 BanI,BshNI,BspT107I,AccB1I
HgiJII 1 Eco24I,EcoT38I,FriOI,BanII
Hin4I 3
Hin4II 4 HpyAV
HindII 1 HincII
HindIII 1
HinfI 5
HpaI 1 KspAI
HpaII 3 HapII,BsiSI,MspI
HphI 2 AsuHPI
Hpy178III 8 Hpy188III
Hpy188I 7
KpnI 1
Ksp632I 1 Eam1104I,EarI,Bst6I
MaeI 9 FspBI,BfaI,XspI
MaeII 2 HpyCH4IV
MaeIII 5
MboI 5 Bsp143I,BssMI,BstMBI,DpnII,Kzo9I,BfuCI,NdeII,Sau3AI
MboII 4
MfeI 2 MunI
MjaIV 5 Hpy8I,Hpy166II
MlyI 1 SchI
MnlI 9
MseI 8 SaqAI,Tru1I,Tru9I
MslI 2 RseI,SmiMI
MwoI 1 HpyF10VI,BstMWI
NdeI 1 FauNDI
NlaIII 7 Hin1II,Hsp92II,FaeI
NlaIV 3 BspLI,BmiI,PspN4I
NspI 1 BstNSI,XceI
PasI 1
PflMI 1 BasI,AccB7I,Van91I
PfoI 1
PleI 1 PpsI
PmaCI 1 BbrPI,Eco72I,AcvI,PmlI,PspCI
RsaI 4 AfaI
SacI 1 Psp124BI,SstI
ScaI 1 BmcAI,AssI,ZrmI
ScrFI 6 BmrFI,MspR9I,Bme1390I
SduI 3 MhlI,Bsp1286I
SecI 6 BseDI,BssECI,BsaJI
SetI 16
SfeI 2 BstSFI,SfcI,BfmI
SpeI 3 BcuI,AhlI
StuI 1 Eco147I,PceI,SseBI,AatI
StyI 2 Eco130I,EcoT14I,ErhI,BssT1I
SwaI 1 SmiI
TaiI 2
TaqI 3
TaqII 1
TatI 1
TfiI 4 PfeI
TscAI 3 TspRI
TseI 3 ApeKI
TsoI 1
Tsp4CI 8 HpyCH4III,TaaI,Bst4CI
TspDTI 10
TspEI 11 TasI,Tsp509I,Sse9I
TstI 1
VspI 1 PshBI,AseI
XbaI 2
XhoII 2 BstYI,MflI,PsuI,BstX2I# Enzymes which cut less frequently than the MINCUTS criterion
# Enzymes < MINCUTS Frequency Isoschizomers# Enzymes which cut more frequently than the MAXCUTS criterion
# Enzymes > MAXCUTS Frequency Isoschizomers# Enzymes that do not cut
AanI AarI AatII AbsI AclI AcyI AflII AgeI
AjuI AlfI AloI ApaI ApaLI ArsI AscI AvaI
AvrII BaeGI BarI BbeI BbvCI Bce83I BcgI BciVI
BclI BdaI BetI BfiI BfoI BglI BglII BmeT110I
BmtI BplI Bpu10I BsaBI BsaXI BsePI BseSI BseYI
BsgI Bsp120I Bsp1407I BspFNI BspLU11I BspMI BspMII BspOI
BsrBI BssNAI Bst1107I BstAFI BstAPI BstEII BstSLI BstZ17I
BtgZI BtrI BtsI Cfr9I CsiI CspCI DinI DraII
DraIII DrdI DsaI Eam1105I EciI Eco47III EcoNI EcoRI
EcoRV EgeI EheI Esp3I FalI FauI FnuDII FseI
FspAI GlaI GsaI HaeII HgaI HhaI Hin6I HinP1I
Hpy99I HspAI KasI KflI MauBI McrI MluI MmeI
MroNI MstI NaeI NarI NcoI NgoMIV NheI NmeAIII
NotI NruI NspBII OliI PacI PmeI PpiI PpuMI
PshAI PsiI PspOMI PspXI PsrI PstI PteI PvuI
PvuII RigI RruI RsrII SacII SalI SanDI SauI
SexAI SfaAI SfiI SfoI SgfI SgrAI SgrDI SmaI
SmlI SnaBI SphI SplI SrfI Sse232I Sse8387I SspDI
SspI TauI Tsp45I TspGWI TspMI Tth111I XcmI XhoI
XmaCI XmaI XmaIII XmnI ZraI
# No. of cutting enzymes which do not match the
# SITELEN, BLUNT, STICKY, COMMERCIAL, AMBIGUOUS citeria781
投稿者 ただ : 23:19 | トラックバック (1) カテゴリー ; 生命科学
2009年5月28日
 マスクのない世界へ
マスクのない世界へ
出張でした。横浜方面まで。…新幹線に乗ったけど(当然か)、マスクしてる人そんなにいなかったね、自分も含め。というか神戸では既にマスクしてJRに乗ってる人も減ったしね。ただ、逆に新大阪の駅では他所からやってくる修学旅行生らしき子達はマスクしてる…なんとも不思議な光景だ。
マスクは感染予防のための道具じゃないんですよ!!
(感染から発症の間までのウィルスの飛散を防ぐには効果があるけど)
まぁ地味ぃに感染地域だけは広がってるね。
主張先には、今回僕は話を聞くだけというなんとも手持ち無沙汰な役柄でしたが、とりあえず話は聞いてきたからそれでいいのだろう。横浜から少し離れた郊外やったけど、何十人かに1人はマスクしてたなぁ…さすがに神戸ほどではないが。
向こうの方でもマスクが品薄らしいけど、なぜか実験用のマスクが在庫切れなのは…なぜだ。医療用に持っていかれたってことか? それとも誰か買い占めたのか? もう高値掴みやと思うぞ。
そしてもう1つ、今回の出張は、ダブルヘッダーでした。…たまの出張だったもんだから、ムリ言ってお願いして…それで19時半まで横浜に居ることになろうとは思いもせんかったけど。毎度のことながら帰ってきたときにはぐったり。
投稿者 ただ : 23:29 | トラックバック (0) カテゴリー ; 仕事関係
2009年5月30日
 ミニマジンガーができるかも
ミニマジンガーができるかも
ニンテンドーDSやAndroidでロボットを操作(動画)(ref. WIREDVISION)
DSがリモコンってのは面白そう。…しかもカメラを通して映像が見える!!
投稿者 ただ : 10:20 | トラックバック (0) カテゴリー ; mein Erbe
2009年5月31日
 スター・トレック
スター・トレック

観に行ってきた。久しぶりに映画館行った気が。やっぱりレ…なんですがね。日本の人たち以外にもGuyたちが多かった、やはり海外にも支持があるだけのことはある。
スタートレックの映画にはいろいろサブタイトル的なものが付いているのしか見たことなかったんやけど、サブタイトルなしって実は初めてなんかな。ファーストコンタクトやらジェネレーションズに近年観たのが全部TNG系やったから、サブタイトルが必須やったのかも知れんけど。いやもっと前にサブタイトルつきのTOSの映画があった気が(うーん、サブタイトルというのかこれは)。
いやいや意外に面白かったね、というか面白いと言う表現以外に表現がないというか、…すんません。話は本流ではないけど、本流とうまくすり合わせているのがなかなか(歴史に変化があっても大局としては進む方向に違いはない、ということ)。何十年も前の時代の作品(の厳密にはカークが船長になるまでの物語)が今になって豪華なCGで構成されてると、不思議な感覚になる。最近のシリーズのCGには違和感ないのになぁ。
未来の自分が自分と話をできるというのは、別宇宙の自分が自分と話しても何もならない、というのが適用されたのか、結局(略歴確認したけど、やっぱり無視されてるし)。
スターゲイト(アトランティス)では当然のように別宇宙から人がやってきたりするから、許されるようになったのかは分からんが。しかしスターゲイトオリジナルでは最終回でティルクはひとこともしゃべらんかった…まぁそれぞれ考えがあってもいい。
いろんなSFの時空連続体に対する観念を考察してみるのも面白いかも。
スールー(もうカトウでない)が意外とさりげなくカッコよかった。
投稿者 ただ : 23:29 | トラックバック (0) カテゴリー ; お出かけ
